最近、Kaggle などのデータ分析コンペで使われることの多い Adversarial Validation という手法について調べたり考えていたので書いてみる。
もくじ
- もくじ
- 背景
- Adversarial Validation
- 試してみる
- 下準備
- 二つのデータが同じ分布に由来するとき
- 二つのデータが異なる分布に由来するとき
- 異なる分布でそのまま分類してみる
- 検証用データに似ているものを取り出す
- 取り出したデータを使って学習してみる
- ラベルごとに似ているデータを取り出す
- どれだけ取り出して学習させれば良いのか
- Adversarial Validation の応用例
背景
Adversarial Validation という手法は、データ分析コンペに存在する、ある課題を解決するために考案された。 その課題とは、提供される複数のデータセットの分布が異なる場合に、いかにして正しく予測するかというもの。
まず、データ分析コンペでは、一般に学習用データと検証用データが提供される。 学習用データには真のラベル情報が与えられるのに対して、検証用データにはそれがない。 参加者は学習用データでトレーニングしたモデルを用いて、検証用データを予測した結果をサブミットする。
サブミットした内容に対するフィードバックは、検証用データの一部を用いて評価した結果として得られる。 検証用データの一部を用いた評価は、Kaggle では Public Leader Board (Public LB) と呼ばれる。 ただし、これはあくまで一部を用いた評価に過ぎないため、検証用データの残りの部分を使ったときにどうなるかは分からない。 検証用データの残りの部分を使った評価は、コンペが終了したときに Private Leader Board (Private LB) として公表される。
Public LB にもとづいてモデルの良し悪しを評価すると、ときとして Private LB で大きく順位が下がることがある。 この現象は、Public LB の評価で用いられる一部のデータに対して、モデルが過剰適合した状態と考えられる。 これを防ぐには、Cross Validation (交差検証) などを用いたローカルでの汎化性能の評価 (Local CV) が重要になる。
しかし、コンペで提供される学習用データと検証用データは、ときとして分布が大きく異なる。 このとき何が起こるかというと、Local CV と Public LB / Private LB のスコアが相関しなくなる。 例えば Local CV では高いスコアが得られるのに、サブミットした結果では低いスコアしか得られない、といった事態が起こる。
Adversarial Validation
この問題を解決するために考案されたのが Adversarial Validation という手法。
Adversarial Validation では、まず二つのデータセットに本来の目的変数とは別の目的変数を設定する。
具体的には、どちらのデータセットに由来するデータなのかという情報を目的変数として付与する。
例えば学習用データが全て 0 で検証用データは全て 1 といった形で付与する。
そして、学習用データと検証用データを混ぜた状態で、両者がどちらに由来するものなのかを予測するモデルを作る。 学習用データと検証用データの分布が異なるほど、どちらに由来するかの予測は容易になる。 もし、全く同じ分布から得られたデータなのであれば、両者を区別することはそもそもできない。 この時点で、学習用データと検証用データの分布がどれくらい異なるのかが分かる。
もし分布が異なるのであれば、学習用データのサブセットを作ることになる。 サブセットを作る上での基準は、検証用データにより近いデータかどうか。 これは、先ほど確認した内容にもとづいて作成する。 つまり、学習用データの中から、検証用データと判断が難しかったものを選び出す。
具体的には、検証用データかどうかの確率を降順ソートして上から取り出せば良い。 その基準で取り出した学習用データのサブセットを使って Local CV することで、Public LB / Private LB との相関が改善する。 以上が Adversarial Validation という手法の説明になる。
試してみる
前置きがだいぶ長くなったけど、ここからは実際に Adversarial Validation を試してみよう。
使った環境は次の通り。
$ sw_vers ProductName: Mac OS X ProductVersion: 10.14.3 BuildVersion: 18D109 $ python -V Python 3.7.2
下準備
まずは、検証に使うパッケージをインストールする。
$ pip install scikit-learn matplotlib
二つのデータが同じ分布に由来するとき
まずは、二つのデータが全く同じ分布に由来するとき Adversarial Validation で何が起こるか確認する。
以下のサンプルコードでは二値分類問題に使うダミーのデータを生成している。
総数 5,000 点の二次元データで、割合は 1:1 の均衡データとなっている。
そのデータを 2,500 点ずつランダムにサンプリングして、これを学習用データと検証用データに模している。
これらのデータに、どちらに由来する要素なのかを表す目的変数 z_* を用意してランダムフォレストで二値分類として解いている。
均衡データなので、評価指標には単純に精度 (Accuracy) を用いた。
#!/usr/bin/env python # -*- coding: utf-8 -*- import numpy as np from sklearn.datasets import make_classification from sklearn.model_selection import train_test_split from sklearn.model_selection import cross_validate from sklearn.model_selection import StratifiedKFold from matplotlib import pyplot as plt from sklearn.ensemble import RandomForestClassifier def main(): # データ点数 5,000 のデータセットを作るパラメータ args = { 'n_samples': 5000, 'n_features': 2, 'n_informative': 2, 'n_redundant': 0, 'n_repeated': 0, 'n_classes': 2, 'n_clusters_per_class': 1, 'flip_y': 0, 'class_sep': 1.5, 'weights': [0.5, 0.5], 'random_state': 42, } # 二次元のデータを生成する X, y = make_classification(**args) # データをランダムに二等分する X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.5, shuffle=True, random_state=42) # 分割したデータがどちらに由来したものか区別するためのラベル z_train = np.zeros(len(X_train)) # 片方は全て 0 z_test = np.ones(len(X_test)) # もう片方は全て 1 # 分割したデータを結合する X_concat = np.concatenate([X_train, X_test], axis=0) z_concat = np.concatenate([z_train, z_test], axis=0) # 上記データをランダムフォレストで分類を試みる clf = RandomForestClassifier(n_estimators=100, random_state=42) # 元々同じ分布から作ったので、どちら由来か分類するのは困難 skf = StratifiedKFold(n_splits=5, shuffle=True, random_state=42) score = cross_validate(clf, X_concat, z_concat, cv=skf) # 5-Fold CV で評価した精度 (Accuracy) の平均 print('Accuracy:', score['test_score'].mean()) # 両データをプロットする plt.scatter(X_train[y_train == 0, 0], X_train[y_train == 0, 1], alpha=0.5, label='Train (Negative)') plt.scatter(X_train[y_train == 1, 0], X_train[y_train == 1, 1], alpha=0.5, label='Train (Positive)') plt.scatter(X_test[y_test == 0, 0], X_test[y_test == 0, 1], alpha=0.5, label='Test (Negative)') plt.scatter(X_test[y_test == 1, 0], X_test[y_test == 1, 1], alpha=0.5, label='Test (Positive)') plt.legend() plt.show() if __name__ == '__main__': main()
上記の実行結果は次の通り。 精度 (Accuracy) は 0.5 を割っており、ランダムに答えたときの期待値よりも低い。
$ python samedist.py
Accuracy: 0.47759999999999997
二つのデータをプロットした散布図は次の通り。
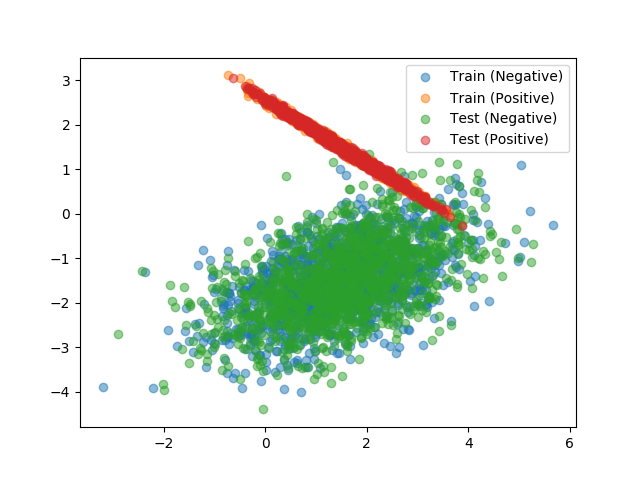
このように、全く同一の分布から得られたものであればデータの由来を予測することは困難といえる。
二つのデータが異なる分布に由来するとき
続いては二つのデータが異なる分布に由来するときに Adversarial Validation で何が起こるかを確認する。
次のサンプルコードでは、乱数を変えて 2,500 点のデータを二つ作っている。 二つのデータはそれなりに似ているものの結構違う、という絶妙なものを探してみた。 先ほどと同じようにどちらのデータに由来するものか目的変数を付与して、それをランダムフォレストで分類している。
#!/usr/bin/env python # -*- coding: utf-8 -*- import numpy as np from sklearn.datasets import make_classification from sklearn.model_selection import cross_validate from sklearn.model_selection import StratifiedKFold from matplotlib import pyplot as plt from sklearn.ensemble import RandomForestClassifier def main(): # データ点数 2,500 のデータセットを作るパラメータ args = { 'n_samples': 2500, 'n_features': 2, 'n_informative': 2, 'n_redundant': 0, 'n_repeated': 0, 'n_classes': 2, 'n_clusters_per_class': 1, 'flip_y': 0, 'class_sep': 1.5, 'weights': [0.5, 0.5], 'random_state': 42, } # 二次元のデータを生成する X_train, y_train = make_classification(**args) # 乱数を変えてもう一度作る args['random_state'] = 424242 X_test, y_test = make_classification(**args) # 分割したデータがどちらに由来したものか区別するためのラベル z_train, z_test = np.zeros(len(X_train)), np.ones(len(X_test)) # 結合する X = np.concatenate([X_train, X_test], axis=0) z = np.concatenate([z_train, z_test], axis=0) # 上記データをランダムフォレストで分類を試みる clf = RandomForestClassifier(n_estimators=100, random_state=42) # 異なる乱数を元にした分布なのでそれなりに分類できる skf = StratifiedKFold(n_splits=5, shuffle=True, random_state=42) score = cross_validate(clf, X, z, cv=skf) print('Accuracy:', score['test_score'].mean()) # 両データをプロットする plt.scatter(X_train[y_train == 0, 0], X_train[y_train == 0, 1], alpha=0.5, label='Train (Negative)') plt.scatter(X_train[y_train == 1, 0], X_train[y_train == 1, 1], alpha=0.5, label='Train (Positive)') plt.scatter(X_test[y_test == 0, 0], X_test[y_test == 0, 1], alpha=0.5, label='Test (Negative)') plt.scatter(X_test[y_test == 1, 0], X_test[y_test == 1, 1], alpha=0.5, label='Test (Positive)') plt.legend() plt.show() if __name__ == '__main__': main()
上記の実行結果は次の通り。
$ python diffdist.py
Accuracy: 0.8498000000000001
今度は、それなりに分類できている。
データをプロットした散布図は次の通り。 Positive なデータの分布が、特に異なることが分かる。
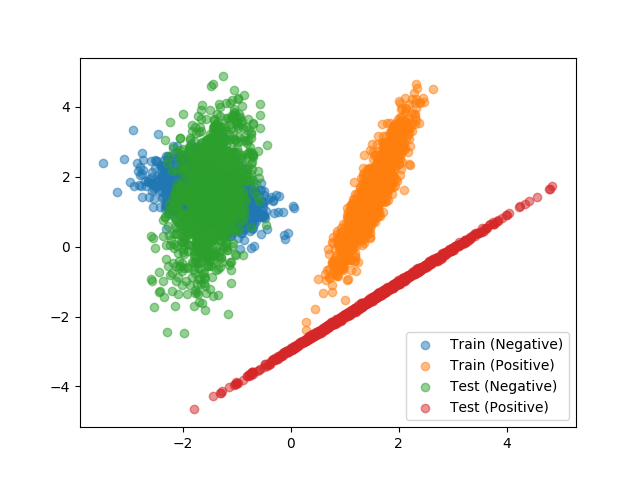
このように、分布が異なる場合には Adversarial Validation で、それを確認できる。
異なる分布でそのまま分類してみる
続いて、先ほど確認した分布の異なるデータを使って学習・予測してみよう。 これは Adversarial Validation を使わないアプローチで結果をサブミットしたときの Private LB のスコアに相当する。
次のサンプルコードでは、先ほど生成したのと同じデータセットを用意した。 そして、学習用データでランダムフォレストをトレーニングしてから、検証用データで予測した場合のスコアを確認している。
#!/usr/bin/env python # -*- coding: utf-8 -*- import numpy as np from matplotlib import pyplot as plt from sklearn.datasets import make_classification from sklearn.metrics import accuracy_score from sklearn.ensemble import RandomForestClassifier def main(): args = { 'n_samples': 2500, 'n_features': 2, 'n_informative': 2, 'n_redundant': 0, 'n_repeated': 0, 'n_classes': 2, 'n_clusters_per_class': 1, 'flip_y': 0, 'class_sep': 1.5, 'weights': [0.5, 0.5], 'random_state': 42, } X_train, y_train = make_classification(**args) args['random_state'] = 424242 X_test, y_test = make_classification(**args) clf = RandomForestClassifier(n_estimators=100, random_state=42) # 最初に作ったデータセットで学習する clf.fit(X_train, y_train) # 次に作ったデータセット (分布が異なる) でモデルを評価する y_pred = clf.predict(X_test) acc = accuracy_score(y_test, y_pred) print('Accuracy:', acc) # モデルの識別境界を可視化する x_mesh, y_mesh = np.meshgrid(np.linspace(-5, 5, 50), np.linspace(-5, 5, 50)) Z = clf.predict(np.c_[x_mesh.ravel(), y_mesh.ravel()]) Z = Z.reshape(x_mesh.shape) plt.contourf(x_mesh, y_mesh, Z, cmap=plt.cm.binary) # 元のデータをプロットする plt.scatter(X_train[y_train == 0, 0], X_train[y_train == 0, 1], alpha=0.5, label='Train (Negative)') plt.scatter(X_train[y_train == 1, 0], X_train[y_train == 1, 1], alpha=0.5, label='Train (Positive)') plt.scatter(X_test[y_test == 0, 0], X_test[y_test == 0, 1], alpha=0.5, label='Test (Negative)') plt.scatter(X_test[y_test == 1, 0], X_test[y_test == 1, 1], alpha=0.5, label='Test (Positive)') plt.legend() plt.show() if __name__ == '__main__': main()
上記の実行結果は次の通り。 精度 (Accuracy) として 0.9516 という結果が得られた。
$ python nochange.py
Accuracy: 0.9516
モデルの識別境界をプロットしたグラフが次の通り。
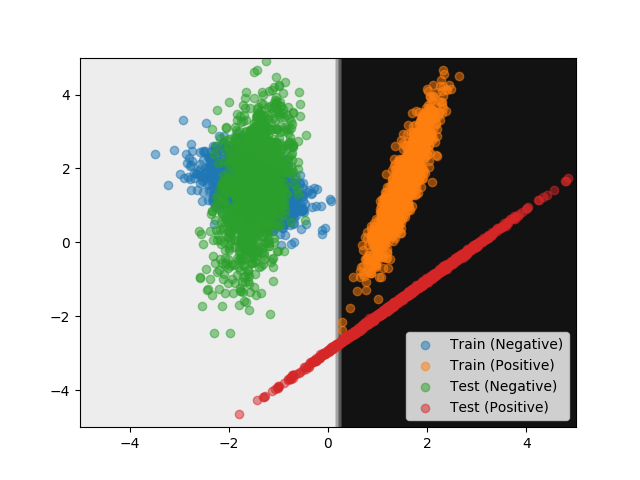
Test (Positive) のデータの一部が誤って分類されていることが分かる。 とはいえモデルは Test に関する情報を知らないので妥当な結果といえる。
検証用データに似ているものを取り出す
続いては、Adversarial Validation を使って検証用データに似ているものを学習用データから取り出してみよう。
次のサンプルコードでは、Adversarial Validation で検証用データに近いと判断された学習用データの上位 1,000 件を取り出している。 ただし、取り出したデータを使った学習まではしておらずグラフへのプロットにとどまる。
#!/usr/bin/env python # -*- coding: utf-8 -*- import numpy as np from sklearn.datasets import make_classification from sklearn.model_selection import cross_val_predict from matplotlib import pyplot as plt from sklearn.ensemble import RandomForestClassifier def main(): args = { 'n_samples': 2500, 'n_features': 2, 'n_informative': 2, 'n_redundant': 0, 'n_repeated': 0, 'n_classes': 2, 'n_clusters_per_class': 1, 'flip_y': 0, 'class_sep': 1.5, 'weights': [0.5, 0.5], 'random_state': 42, } X_train, y_train = make_classification(**args) args['random_state'] = 424242 X_test, y_test = make_classification(**args) # 分割したデータがどちらに由来したものか区別するためのラベル z_train, z_test = np.zeros(len(X_train)), np.ones(len(X_test)) X = np.concatenate([X_train, X_test], axis=0) z = np.concatenate([z_train, z_test], axis=0) # 要素が最初のデータセット由来なのか、次のデータセット由来なのか分類する clf = RandomForestClassifier(n_estimators=100, random_state=42) # 分類して確率を計算する z_pred_proba = cross_val_predict(clf, X, z, cv=5, method='predict_proba') # 先に生成したデータに対応する部分だけ取り出す z_train_pred_proba = z_pred_proba[:2500] # 後から作ったデータに近いと判断されたものを取り出す X_train_alike = X_train[np.argsort(z_train_pred_proba[:, 0])][:1000] # 上記の残り X_train_rest = X_train[np.argsort(z_train_pred_proba[:, 0])][1000:] # それぞれのデータをプロットする plt.scatter(X_test[:, 0], X_test[:, 1], alpha=0.5, label='Test') plt.scatter(X_train_rest[:, 0], X_train_rest[:, 1], alpha=0.5, label='NOT Resemblance') plt.scatter(X_train_alike[:, 0], X_train_alike[:, 1], alpha=0.5, label='Resemblance') plt.grid() plt.legend() plt.show() if __name__ == '__main__': main()
上記を実行してみる。
$ python resemblance.py
得られるグラフは次の通り。
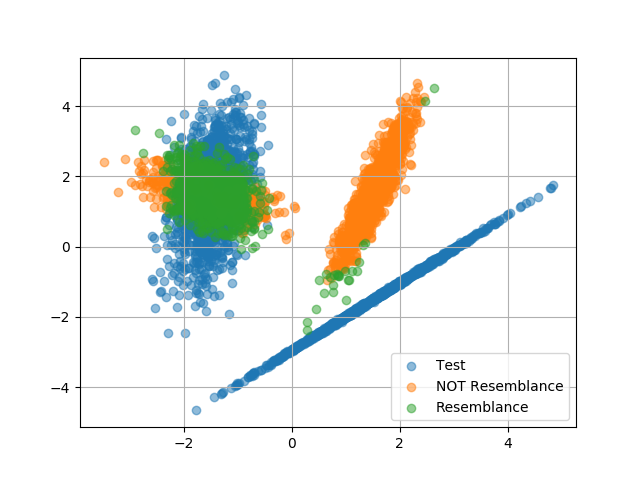
Negative なデータにおいては中心部分が、Positive なデータにおいてはエッジ部分が似ていると判断されたようだ。
取り出したデータを使って学習してみる
続いては、先ほど取り出したデータを使って実際に学習してみよう。
以下のサンプルコードでは、先ほど取り出したのと同じデータを使ってモデルを学習している。 つまり、検証用データに近いと判断された学習用データの要素 1,000 点を使った。 そして、学習させたモデルを使って検証用データのスコアを計算している。 また、同時にモデルの識別境界も可視化した。
#!/usr/bin/env python # -*- coding: utf-8 -*- import numpy as np from matplotlib import pyplot as plt from sklearn.datasets import make_classification from sklearn.metrics import accuracy_score from sklearn.model_selection import cross_val_predict from sklearn.ensemble import RandomForestClassifier def main(): args = { 'n_samples': 2500, 'n_features': 2, 'n_informative': 2, 'n_redundant': 0, 'n_repeated': 0, 'n_classes': 2, 'n_clusters_per_class': 1, 'flip_y': 0, 'class_sep': 1.5, 'weights': [0.5, 0.5], 'random_state': 42, } X_train, y_train = make_classification(**args) args['random_state'] = 424242 X_test, y_test = make_classification(**args) z_train, z_test = np.zeros(len(X_train)), np.ones(len(X_test)) X = np.concatenate([X_train, X_test], axis=0) z = np.concatenate([z_train, z_test], axis=0) # 要素が最初のデータセット由来なのか、次のデータセット由来なのか分類する clf = RandomForestClassifier(n_estimators=100, random_state=42) z_pred_proba = cross_val_predict(clf, X, z, cv=5, method='predict_proba') # 先に生成したデータに対応する部分だけ取り出す z_train_pred_proba = z_pred_proba[:2500] # 後から作ったデータに近いと判断されたものを取り出す X_train_alike = X_train[np.argsort(z_train_pred_proba[:, 0])][:1000] y_train_alike = y_train[np.argsort(z_train_pred_proba[:, 0])][:1000] # 似ているデータで学習する clf.fit(X_train_alike, y_train_alike) # 後から作ったデータで評価する y_pred = clf.predict(X_test) # 精度 (Accuracy) を確認する acc = accuracy_score(y_test, y_pred) print('Accuracy:', acc) # 識別境界を可視化する x_mesh, y_mesh = np.meshgrid(np.linspace(-5, 5, 50), np.linspace(-5, 5, 50)) Z = clf.predict(np.c_[x_mesh.ravel(), y_mesh.ravel()]) Z = Z.reshape(x_mesh.shape) plt.contourf(x_mesh, y_mesh, Z, cmap=plt.cm.binary) plt.scatter(X_train[y_train == 0, 0], X_train[y_train == 0, 1], alpha=0.5, label='Train (Negative)') plt.scatter(X_train[y_train == 1, 0], X_train[y_train == 1, 1], alpha=0.5, label='Train (Positive)') plt.scatter(X_test[y_test == 0, 0], X_test[y_test == 0, 1], alpha=0.5, label='Test (Negative)') plt.scatter(X_test[y_test == 1, 0], X_test[y_test == 1, 1], alpha=0.5, label='Test (Positive)') plt.scatter(X_train_alike[:, 0], X_train_alike[:, 1], label='Model Learned') plt.legend() plt.show() if __name__ == '__main__': main()
上記の実行結果は次の通り。 先ほど、学習用データをそのまま使ったパターンよりもスコアが改善している。
$ python advlearn.py
Accuracy: 0.9788
識別境界を示すグラフは次の通り。
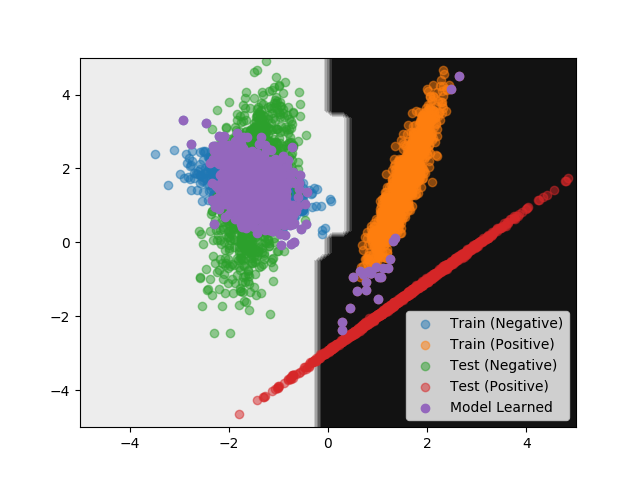
ラベルごとに似ているデータを取り出す
先ほどの例ではラベルによらず検証用データに似たものを学習用データから 1,000 件取り出した。 ただ、可視化した内容を見ても分かる通り、似ているかどうかはラベルによって異なる場合がある。 例えば、先ほどの例では Negative なラベルに、より多くの似ているデータが含まれていた。 これでは、モデルに学習させるラベルの数が不均衡になる問題点がある。 なので、続いてはラベルごとに似ているデータを取り出してみることにしよう。
次のサンプルコードでは、ラベルごとに似ているデータを 200 件ずつ取り出していて学習に使っている。 取り出したデータの可視化と識別境界をプロットしている点については先ほどと変わらない。
#!/usr/bin/env python # -*- coding: utf-8 -*- import numpy as np from matplotlib import pyplot as plt from sklearn.datasets import make_classification from sklearn.metrics import accuracy_score from sklearn.model_selection import cross_val_predict from sklearn.ensemble import RandomForestClassifier def main(): args = { 'n_samples': 2500, 'n_features': 2, 'n_informative': 2, 'n_redundant': 0, 'n_repeated': 0, 'n_classes': 2, 'n_clusters_per_class': 1, 'flip_y': 0, 'class_sep': 1.5, 'weights': [0.5, 0.5], 'random_state': 42, } X_train, y_train = make_classification(**args) args['random_state'] = 424242 X_test, y_test = make_classification(**args) z_train, z_test = np.zeros(len(X_train)), np.ones(len(X_test)) X = np.concatenate([X_train, X_test], axis=0) z = np.concatenate([z_train, z_test], axis=0) # 要素が最初のデータセット由来なのか、次のデータセット由来なのか分類する clf = RandomForestClassifier(n_estimators=100, random_state=42) z_pred_proba = cross_val_predict(clf, X, z, cv=5, method='predict_proba') # 先に生成したデータに対応する部分だけ取り出す z_train_pred_proba = z_pred_proba[:2500] # 後から生成したデータへの近さにもとづいてソートする X_train_alike = X_train[np.argsort(z_train_pred_proba[:, 0])] y_train_alike = y_train[np.argsort(z_train_pred_proba[:, 0])] # 近いものをラベルごとに取り出す X_train_alike_neg = X_train_alike[y_train_alike == 0][:100] y_train_alike_neg = y_train_alike[y_train_alike == 0][:100] X_train_alike_pos = X_train_alike[y_train_alike == 1][:100] y_train_alike_pos = y_train_alike[y_train_alike == 1][:100] X_train_alike_concat = np.concatenate([X_train_alike_neg, X_train_alike_pos], axis=0) y_train_alike_concat = np.concatenate([y_train_alike_neg, y_train_alike_pos], axis=0) # 似ているデータで学習する clf.fit(X_train_alike_concat, y_train_alike_concat) # 後から作ったデータで評価する y_pred = clf.predict(X_test) # 精度 (Accuracy) を確認する acc = accuracy_score(y_test, y_pred) print('Accuracy:', acc) # 識別境界を可視化する x_mesh, y_mesh = np.meshgrid(np.linspace(-5, 5, 50), np.linspace(-5, 5, 50)) Z = clf.predict(np.c_[x_mesh.ravel(), y_mesh.ravel()]) Z = Z.reshape(x_mesh.shape) plt.contourf(x_mesh, y_mesh, Z, cmap=plt.cm.binary) plt.scatter(X_train[y_train == 0, 0], X_train[y_train == 0, 1], alpha=0.5, label='Train (Negative)') plt.scatter(X_train[y_train == 1, 0], X_train[y_train == 1, 1], alpha=0.5, label='Train (Positive)') plt.scatter(X_test[y_test == 0, 0], X_test[y_test == 0, 1], alpha=0.5, label='Test (Negative)') plt.scatter(X_test[y_test == 1, 0], X_test[y_test == 1, 1], alpha=0.5, label='Test (Positive)') plt.scatter(X_train_alike_concat[:, 0], X_train_alike_concat[:, 1], label='Model Learned') plt.legend() plt.show() if __name__ == '__main__': main()
上記の実行結果は次の通り。 先ほどよりもスコアがさらに改善している。
$ python perlabel.py
Accuracy: 0.9812
取り出したデータと識別境界をプロットしたグラフは次の通り。
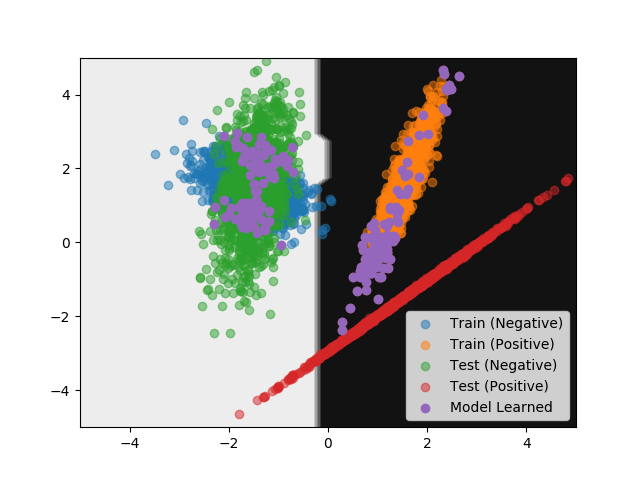
どれだけ取り出して学習させれば良いのか
ここまでで、検証用データに似たデータを学習用データから取り出して使うとスコアが上昇する可能性があることが分かった。 とはいえ、まだ疑問が残っている。 一体、どれだけ取り出して学習させると良い結果が得られるのだろうか。 先に断っておくと、まだ自分の中で結論は出ていない。 先ほど用いた上位 200 件というのは、特に根拠のない適当に選んだ数値だった。
試しに次のサンプルコードでは取り出す件数と精度の推移をプロットしている。 また、そのとき選ばれた末尾のデータは Adversarial Validation でどれくらい「検証用データっぽい」と判断されたのかも可視化した。
#!/usr/bin/env python # -*- coding: utf-8 -*- import numpy as np from matplotlib import pyplot as plt from sklearn.datasets import make_classification from sklearn.metrics import accuracy_score from sklearn.model_selection import cross_val_predict from sklearn.ensemble import RandomForestClassifier def main(): args = { 'n_samples': 2500, 'n_features': 2, 'n_informative': 2, 'n_redundant': 0, 'n_repeated': 0, 'n_classes': 2, 'n_clusters_per_class': 1, 'flip_y': 0, 'class_sep': 1.5, 'weights': [0.5, 0.5], 'random_state': 42, } X_train, y_train = make_classification(**args) args['random_state'] = 424242 X_test, y_test = make_classification(**args) z_train, z_test = np.zeros(len(X_train)), np.ones(len(X_test)) X = np.concatenate([X_train, X_test], axis=0) z = np.concatenate([z_train, z_test], axis=0) # 要素が最初のデータセット由来なのか、次のデータセット由来なのか分類する clf = RandomForestClassifier(n_estimators=100, random_state=42) z_pred_proba = cross_val_predict(clf, X, z, cv=5, method='predict_proba') # 先に生成したデータに対応する部分だけ取り出す z_train_pred_proba = z_pred_proba[:2500] # 後から生成したデータへの近さにもとづいてソートする sorted_train_index_test_similarity = np.argsort(z_train_pred_proba[:, 0]) X_train_alike = X_train[sorted_train_index_test_similarity] y_train_alike = y_train[sorted_train_index_test_similarity] sorted_train_proba_test_similarity = np.sort(z_train_pred_proba[:, 0]) accuracies = [] probabilities = [] picks = list(range(100, 2500, 100)) for i in picks: # 近いものをラベルごとに取り出す X_train_alike_neg = X_train_alike[y_train_alike == 0][:i] y_train_alike_neg = y_train_alike[y_train_alike == 0][:i] X_train_alike_pos = X_train_alike[y_train_alike == 1][:i] y_train_alike_pos = y_train_alike[y_train_alike == 1][:i] X_train_alike_concat = np.concatenate([X_train_alike_neg, X_train_alike_pos], axis=0) y_train_alike_concat = np.concatenate([y_train_alike_neg, y_train_alike_pos], axis=0) # 似ているデータで学習する clf.fit(X_train_alike_concat, y_train_alike_concat) # 後から作ったデータで評価する y_pred = clf.predict(X_test) # 精度 (Accuracy) を確認する acc = accuracy_score(y_test, y_pred) accuracies.append(acc) # 取り出したデータの検証用データっぽさ (確率) を確認する proba = 1.0 - sorted_train_proba_test_similarity[i] probabilities.append(proba) # 結果をプロットする _, ax1 = plt.subplots(figsize=(8, 4)) ax1.plot(picks, accuracies, c='b') ax1.set_xlabel('picks') ax1.set_ylabel('accuracy') ax2 = ax1.twinx() ax2.plot(picks, probabilities, c='r') ax2.set_xlabel('picks') ax2.set_ylabel('probability') plt.show() if __name__ == '__main__': main()
なお、この精度の推移は Private LB の結果と同じ意味を持っているので、コンペ開催中にこのような探索をすることはできない。 代わりに、Public LB であれば Local CV との相関の推移を比較することはできると思う。
上記を実行してみる。
$ python pickacc.py
得られたグラフは次の通り。
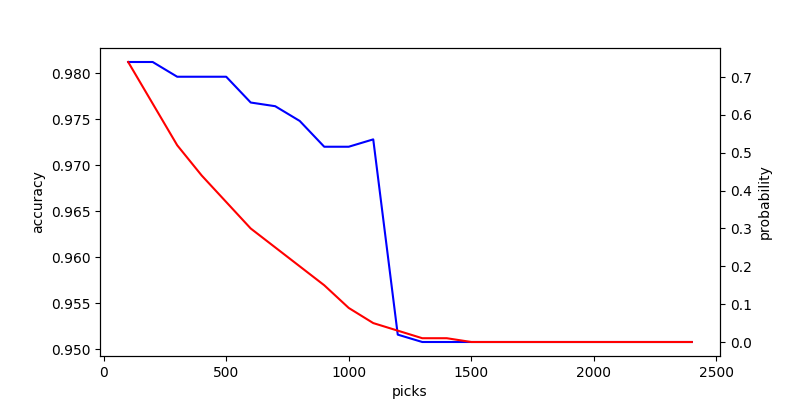
どうやら、今回のデータでは上位 1,200 件を取り出すあたりから精度が大きく改善していることが分かる。 そして、結局上位 100 ~ 200 件を取り出すのが、最も良い結果が得られていた。 今回のケースでは、学習用データを大きく捨ててでも、より似ているデータを取り出す方が良い結果になるようだ。
ただし、上記は用いるデータによって結果は異なると考えられる。 おそらく Adversarial Validation でどれだけ高いスコアが得られたか、つまり分布が似ているか否かで取り出す量を変化させるのが良い気がしている。
Adversarial Validation の応用例
主にデータ分析コンペで用いられる Adversarial Validation だけど、おそらく現実世界でも応用が効くところはあると考えている。
例えば、運用中の機械学習モデルの再学習をするタイミングの判断に使えたりするんじゃないだろうか。 このアイデアでは、モデルの学習に使ったデータと、リアルタイムのデータを Adversarial Validation する。 モデルの学習に使ったデータとリアルタイムのデータの分布が近ければ、分類はできないはず。 しかし、分布が異なってくれば分類できるようになる。 この場合、運用中の機械学習モデルは未知の分布を処理していることになるので性能が低下している恐れがある。
このように、本来の目的変数とは異なる目的変数をデータに付与して分類させるという Adversarial Validation の考え方は、色々と応用が効きそうな気がしている。
いじょう。

スマートPythonプログラミング: Pythonのより良い書き方を学ぶ
- 作者:もみじあめ
- 発売日: 2016/03/12
- メディア: Kindle版