今回は、Python の有名な可視化ライブラリである matplotlib のラッパーとして動作する seaborn を試してみる。
seaborn を使うと、よく必要になる割に matplotlib をそのまま使うと面倒なグラフが簡単に描ける。
毎回、使うときに検索することになるので備忘録を兼ねて。
使った環境は次の通り。
$ sw_vers
ProductName: Mac OS X
ProductVersion: 10.14.4
BuildVersion: 18E226
$ python -V
Python 3.7.3
もくじ
下準備
下準備として seaborn をインストールしておく。
$ pip install seaborn
今回は Python のインタプリタ上で動作確認する。
$ python
まずは seaborn と matplotlib をインポートする。
>>> import seaborn as sns
>>> from matplotlib import pyplot as plt
グラフが見やすいようにスタイルを設定する。
>>> sns.set(style='darkgrid')
Relational plots
まずは seaborn の中で「Relational plots」というカテゴリに属するグラフから試していく。
scatter plot (散布図)
まずは散布図から。
動作確認のために "tips" という名前のデータセットを読み込む。
これは、レストランでの支払いに関するデータセットになっている。
>>> tips = sns.load_dataset('tips')
>>> type(tips)
<class 'pandas.core.frame.DataFrame'>
>>> tips.head()
total_bill tip sex smoker day time size
0 16.99 1.01 Female No Sun Dinner 2
1 10.34 1.66 Male No Sun Dinner 3
2 21.01 3.50 Male No Sun Dinner 3
3 23.68 3.31 Male No Sun Dinner 2
4 24.59 3.61 Female No Sun Dinner 4
散布図を描くときは scatterplot() という関数を使う。
>>> sns.scatterplot(data=tips, x='total_bill', y='tip')
<matplotlib.axes._subplots.AxesSubplot object at 0x1166db390>
関数を呼び出したら pyplot.show() 関数を実行しよう。
なお、以降は plt.show() の実行については省略する。
>>> plt.show()
すると、次のようなグラフが得られる。
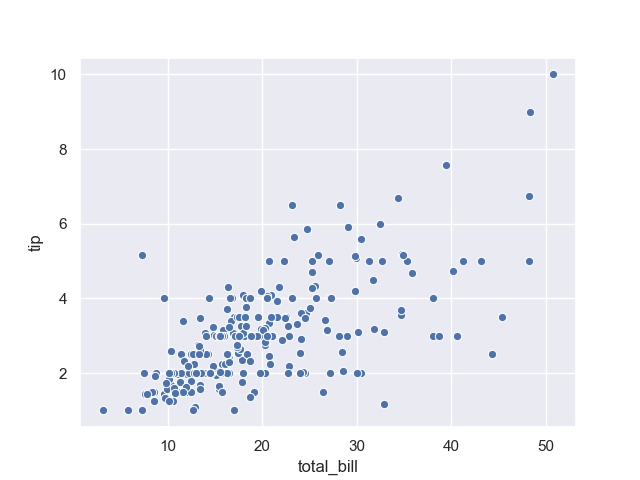
上記では、支払い総額とチップの関係性を散布図で可視化している。
それなりに相関がありそうだ。
続いては、喫煙者と非喫煙者で傾向に差があるかどうか見てみよう。
一つのグラフの中で見比べるときは hue オプションを使うと良い。
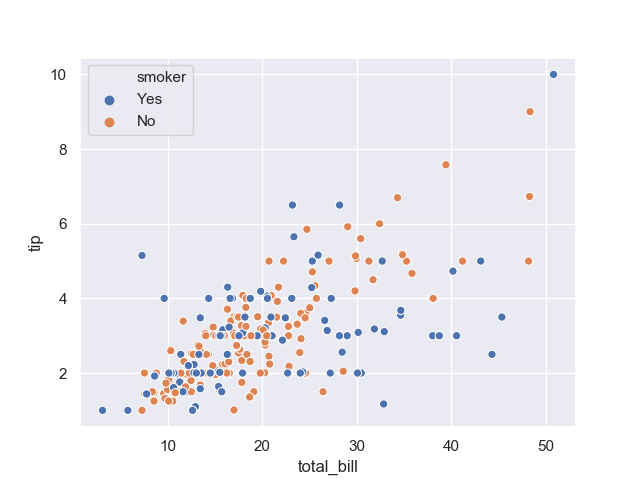
>>> sns.scatterplot(data=tips, x='total_bill', y='tip', hue='smoker')
<matplotlib.axes._subplots.AxesSubplot object at 0x11e1949b0>
以下のようなグラフが得られる。
hue オプション以外にも、一つのグラフの中で違うことを示すには style や size といったオプションも使える。
例えば style を指定してみよう。
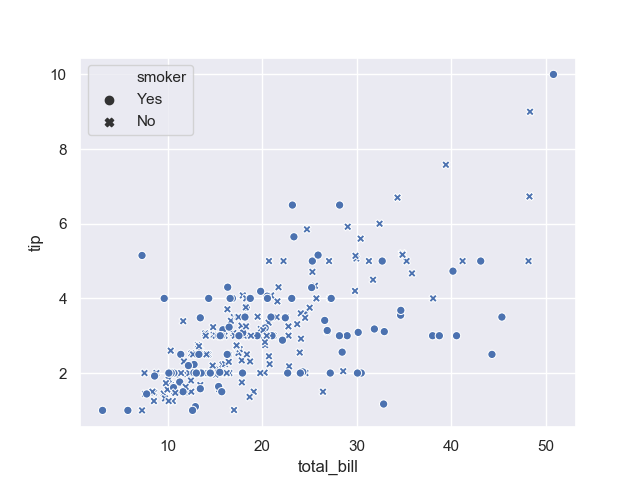
>>> sns.scatterplot(data=tips, x='total_bill', y='tip', style='smoker')
<matplotlib.axes._subplots.AxesSubplot object at 0x11c43e588>
すると、次のようにマーカーの形が変わる。
同様に size を指定してみる。
>>> sns.scatterplot(data=tips, x='total_bill', y='tip', size='smoker')
<matplotlib.axes._subplots.AxesSubplot object at 0x11c4a2908>
すると、次のようにマーカーの大きさが変わる。
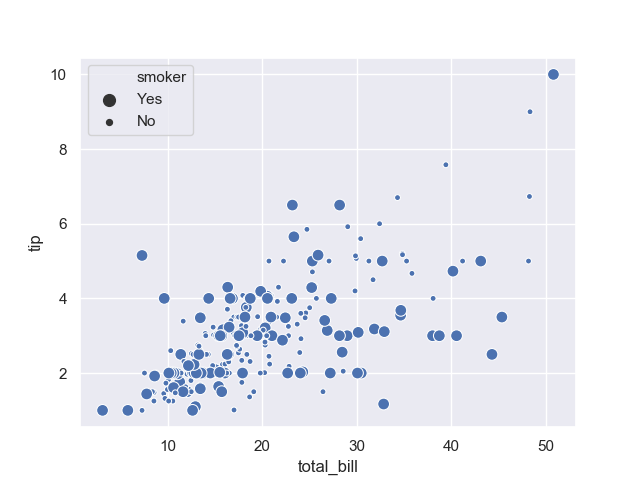
もちろん、これらのオプションは混ぜて使うこともできる。
例えば喫煙者か非喫煙者か以外に、性別や時間 (ランチ・ディナー) について指定してみよう。
>>> sns.scatterplot(data=tips, x='total_bill', y='tip', hue='smoker', style='sex', size='time')
<matplotlib.axes._subplots.AxesSubplot object at 0x11da1a400>
次のようなグラフが得られる。
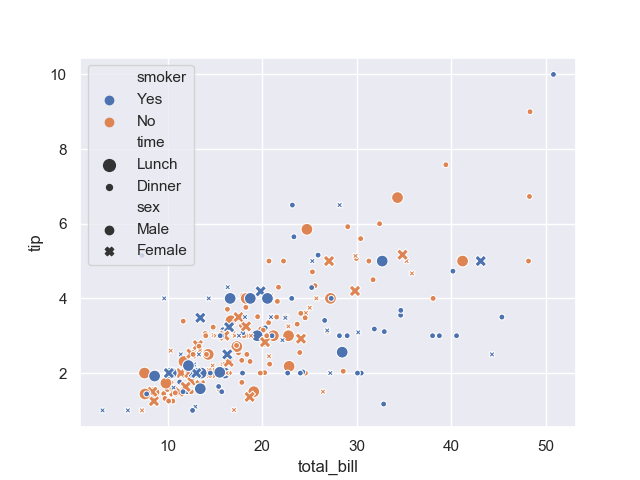
うん、まったく訳がわからない。
一つのグラフには情報を詰め込みすぎないように気をつけよう。
なお、ここまで使ってきた hue や style や size といったオプションは別の API でも使える場合が多い。
また、relplot() 関数を使うと複数の散布図を扱うことができる。
relplot() 関数は scatterplot() 関数を、より一般化した API となっている。
散布図は kind='scatter' と指定することで扱える。
同時に col オプションを指定すると、そこに指定したカラムごとに別々のグラフが得られる。
>>> sns.relplot(data=tips, kind='scatter', x='total_bill', y='tip', col='smoker')
<seaborn.axisgrid.FacetGrid object at 0x102e0b0f0>
上記で得られるグラフが以下。
喫煙者は支払総額とチップの相関が非喫煙者に比べるとやや低いように見受けられる。
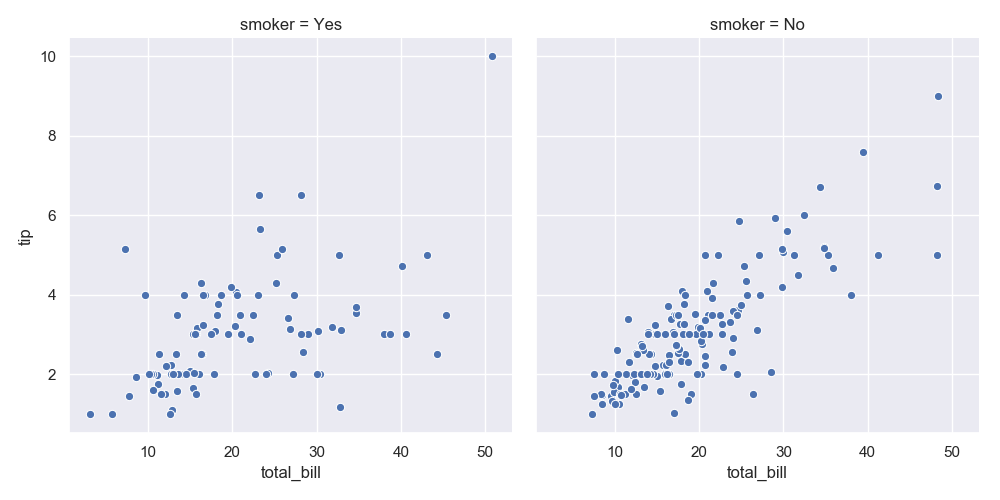
実際に確認してみよう。
>>> tips.corr()['total_bill']['tip']
0.6757341092113642
>>> tips[tips.smoker == 'No'].corr()['total_bill']['tip']
0.822182625705083
>>> tips[tips.smoker == 'Yes'].corr()['total_bill']['tip']
0.4882179411628103
全体では相関係数が 0.675 だったのに対して非喫煙者で層化すると 0.822 となり喫煙者では 0.488 となった。
複数のグラフに分割すると、情報を詰め込みすぎて見にくいグラフになることを防げる。
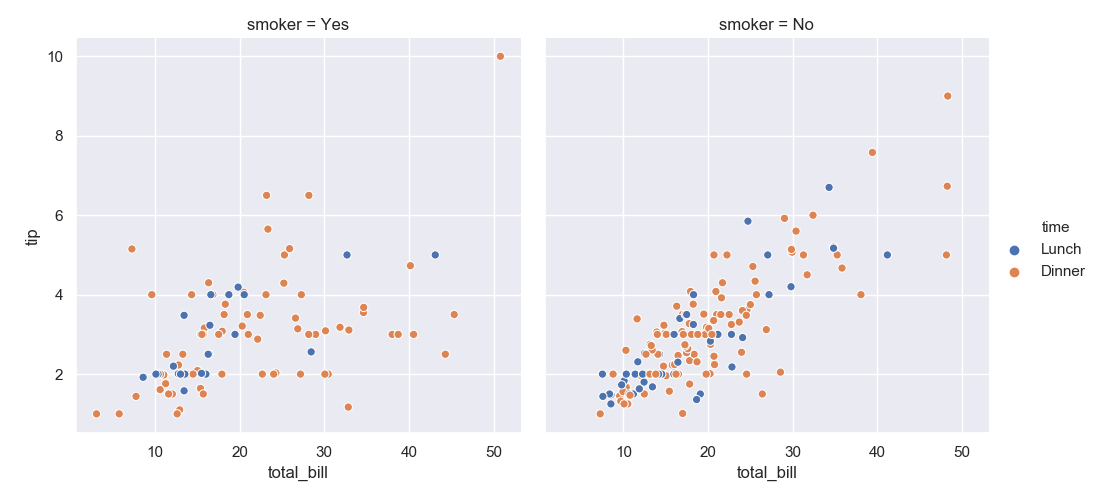
試しに col と hue を併用してみよう。
>>> sns.relplot(data=tips, kind='scatter', x='total_bill', y='tip', hue='time', col='smoker')
<seaborn.axisgrid.FacetGrid object at 0x11e5ee470>
以下のグラフでは喫煙者・非喫煙者でグラフを分けつつ、各グラフの中では時間によるチップ額の傾向を分けて示している。
line plot (折れ線グラフ)
続いては折れ線グラフを試す。
動作確認のために "flights" というデータセットを読み込もう。
これは、飛行機の乗客数の推移を示している。
>>> flights = sns.load_dataset('flights')
>>> flights.head()
year month passengers
0 1949 January 112
1 1949 February 118
2 1949 March 132
3 1949 April 129
4 1949 May 121
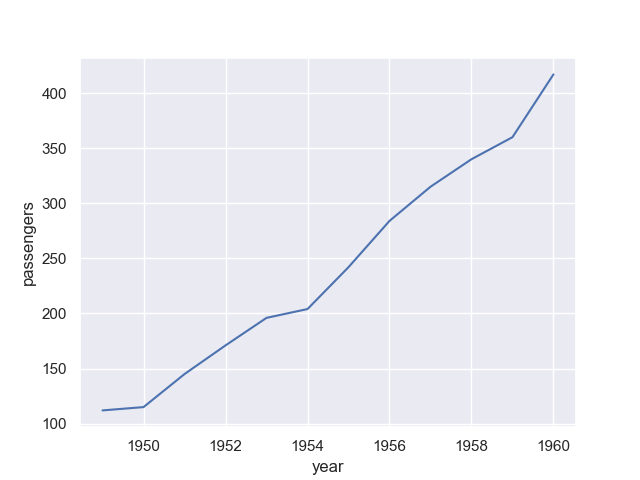
試しに 1 月の乗客の推移を年ごとに可視化してみよう。
折れ線グラフの描画には lineplot() 関数を使う。
>>> sns.lineplot(data=flights[flights.month == 'January'], x='year', y='passengers')
<matplotlib.axes._subplots.AxesSubplot object at 0x11c6c6b00>
上記から得られるグラフは次の通り。
乗客の数は右肩上がりのようだ。
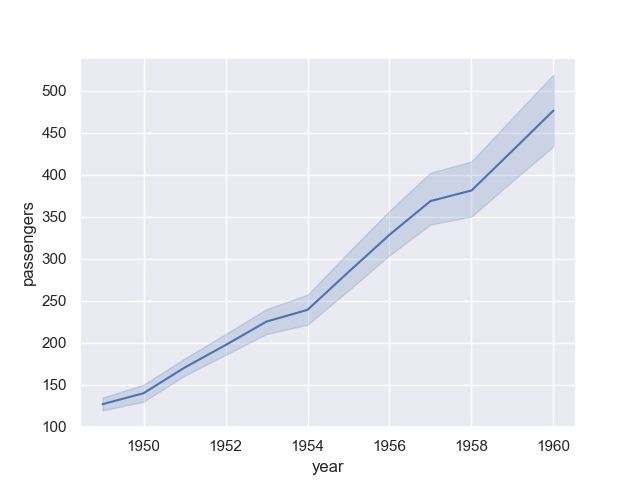
特定の月に限定しない場合についても確認しておこう。
>>> sns.lineplot(data=flights, x='year', y='passengers')
<matplotlib.axes._subplots.AxesSubplot object at 0x11e659940>
上記から得られたグラフが次の通り。
今度は実線の上下に範囲を指定するようなグラフになった。
これはデフォルトではブートストラップ信頼区間 (信頼係数 95%)を示しているらしい。
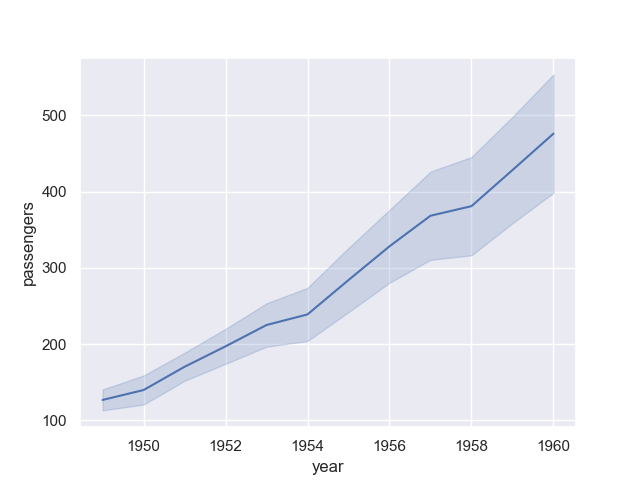
ci オプションに sd を指定することで、標準偏差を用いた信頼区間にもできるようだ。
使うのは、分散が正規分布と仮定できる場合?
>>> sns.lineplot(data=flights, x='year', y='passengers', ci='sd')
<matplotlib.axes._subplots.AxesSubplot object at 0x11e69bb00>
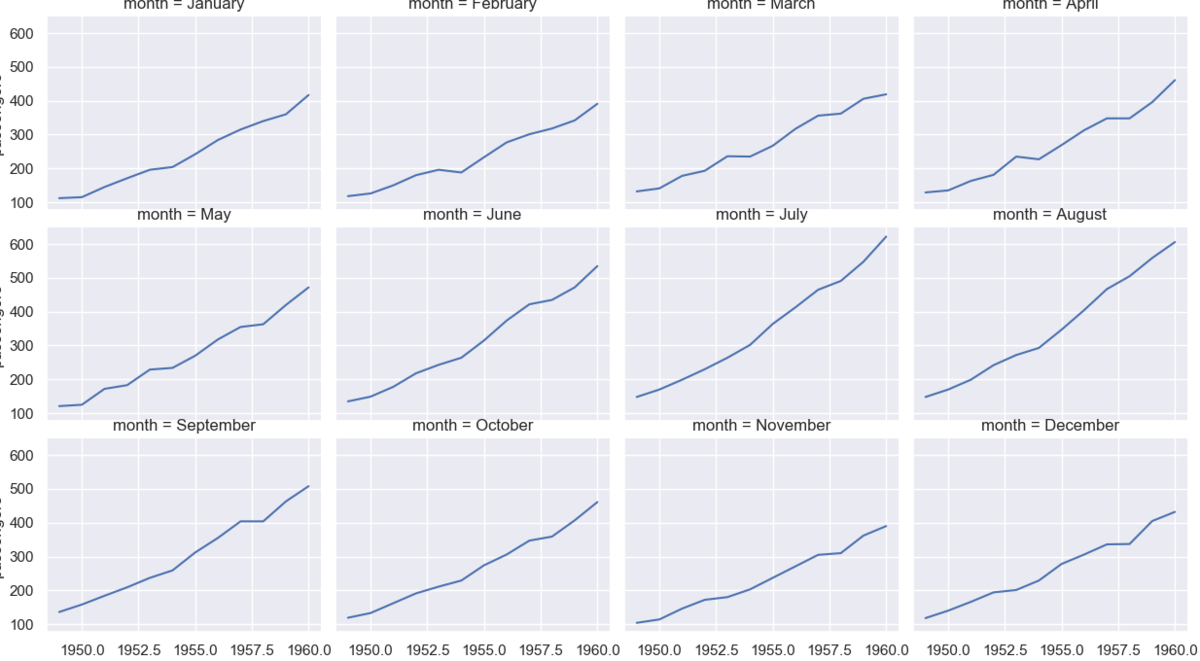
複数のグラフに分けて表示したいときは scatterplot() のときと同じように relplot() を使う。
ただし、kind には line を指定する。
また、数が多いときは横に並んでしまうので col_wrap を指定することで折り返すと良い。
>>> sns.relplot(data=flights, kind='line', x='year', y='passengers', col='month', col_wrap=4)
<seaborn.axisgrid.FacetGrid object at 0x11e631898>
Categorical plots
続いては "Categorical plots" に分類されるグラフを見ていく。
動作確認のために "titanic" データセットを読み込む。
タイタニック号の沈没に関する乗客のデータセット。
>>> titanic = sns.load_dataset('titanic')
>>> titanic.head()
survived pclass sex age sibsp parch ... who adult_male deck embark_town alive alone
0 0 3 male 22.0 1 0 ... man True NaN Southampton no False
1 1 1 female 38.0 1 0 ... woman False C Cherbourg yes False
2 1 3 female 26.0 0 0 ... woman False NaN Southampton yes True
3 1 1 female 35.0 1 0 ... woman False C Southampton yes False
4 0 3 male 35.0 0 0 ... man True NaN Southampton no True
[5 rows x 15 columns]
strip plot (ストリップチャート)
まずはストリップチャートから。
客室のグレードと年齢の関係性についてプロットしてみよう。
>>> sns.stripplot(data=titanic, x='pclass', y='age')
<matplotlib.axes._subplots.AxesSubplot object at 0x11d434748>
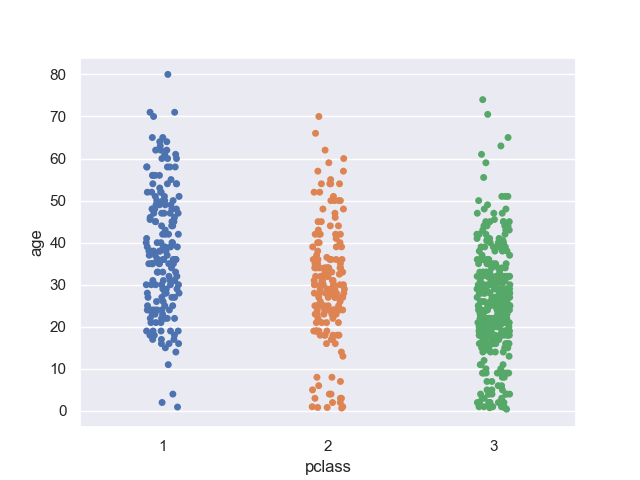
客室のグレードが高い方が年齢層が高め。
性別で層化してみる。
>>> sns.stripplot(data=titanic, x='pclass', y='age', hue='sex')
<matplotlib.axes._subplots.AxesSubplot object at 0x11d58d6d8>
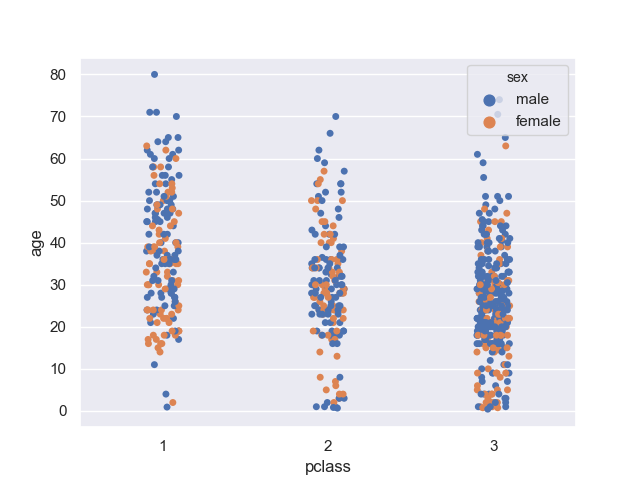
混ざってしまって見にくいときは dodge オプションを True にすると良い。
>>> sns.stripplot(data=titanic, x='pclass', y='age', hue='sex', dodge=True)
<matplotlib.axes._subplots.AxesSubplot object at 0x11d48ca20>
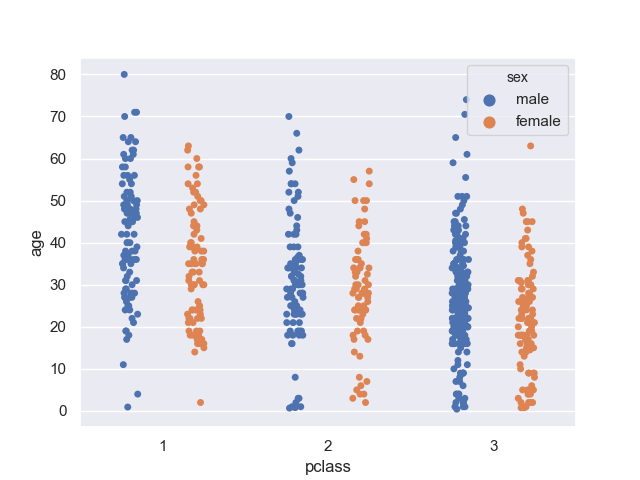
女性の方が、やや年齢層が低そう?
家族など、男性と一緒に来ている影響もあるだろうか。
生死で層化した場合についても見てみよう。
複数のグラフに分けたいときは catplot() 関数を使う。
その際、kind オプションには strip を指定する。
これは scatterplot() や lineplot() で複数のグラフを扱うときに relplot() を使ったのと同じ考え方。
>>> sns.catplot(data=titanic, kind='strip', x='pclass', y='age', hue='survived', col='sex', dodge=True)
<seaborn.axisgrid.FacetGrid object at 0x11d47a4a8>
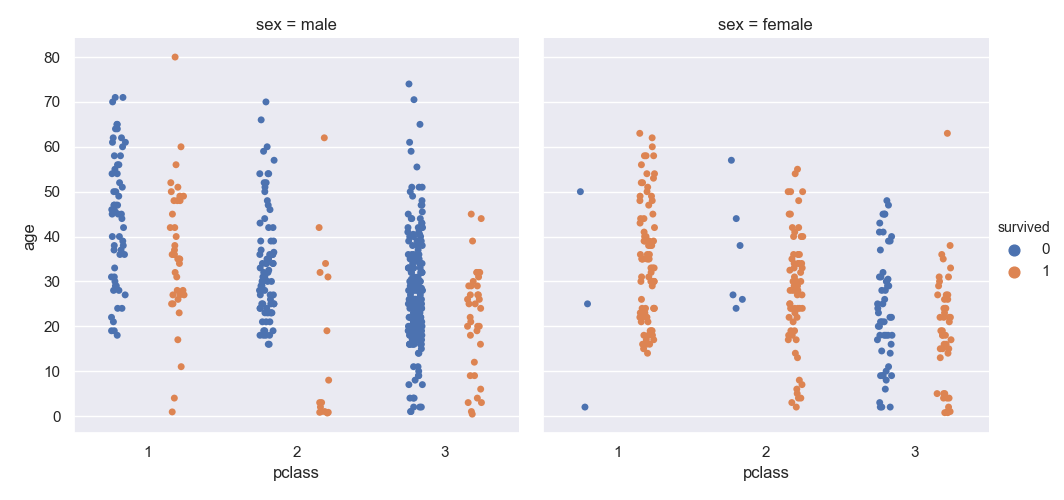
あきらかに、一等客室と二等客室の女性は生き残りやすかったことが分かる。
swarm plot (スウォームチャート)
ストリップチャートは要素が重なっていたけど、重なりを除外したものがこちら。
swarmplot() 関数を使うことで描画できる。
>>> sns.swarmplot(data=titanic, x='pclass', y='age')
<matplotlib.axes._subplots.AxesSubplot object at 0x11daa6320>
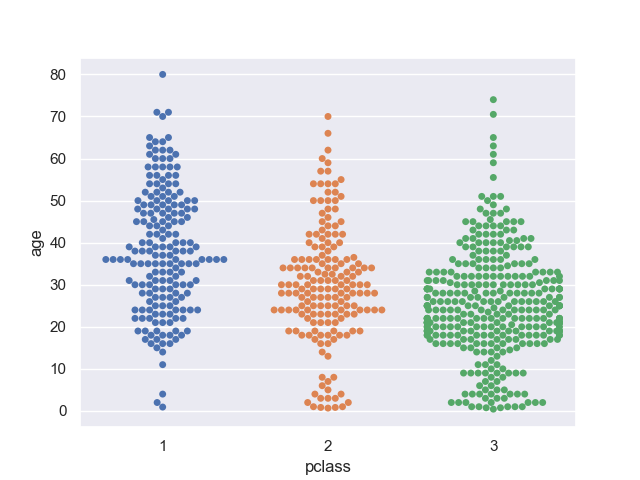
似たような値の数がどれくらいあるかは分かりやすいかも。
box plot (箱ひげ図)
これは多くの人に馴染みがあると思う。
箱ひげ図は boxplot() 関数を使って描画する。
>>> sns.boxplot(data=titanic, x='pclass', y='age')
<matplotlib.axes._subplots.AxesSubplot object at 0x11d5bc7b8>
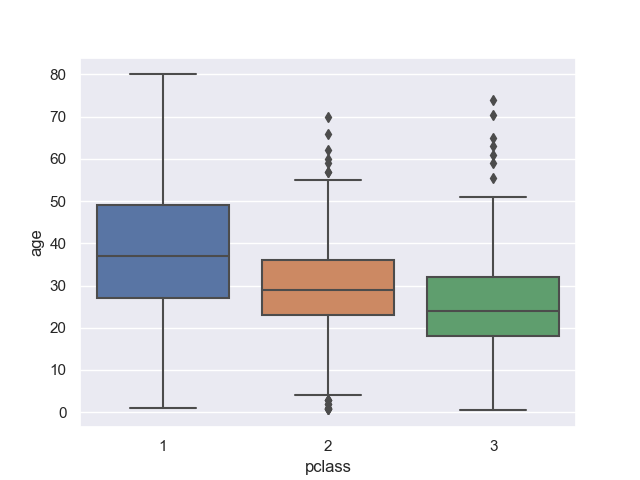
最大値、第二四分位数、中央値、第三四分位数、最小値、外れ値を確認できる。
外れ値は第二、第三四分位数から 1.5 IQR (Interquartile Range) の外にあるものになる。
複数のグラフに分けて表示したいときは catplot() を使いつつ kind オプションに box を指定する。
>>> sns.catplot(data=titanic, kind='box', x='pclass', y='age', hue='survived', col='sex')
<seaborn.axisgrid.FacetGrid object at 0x11da78588>
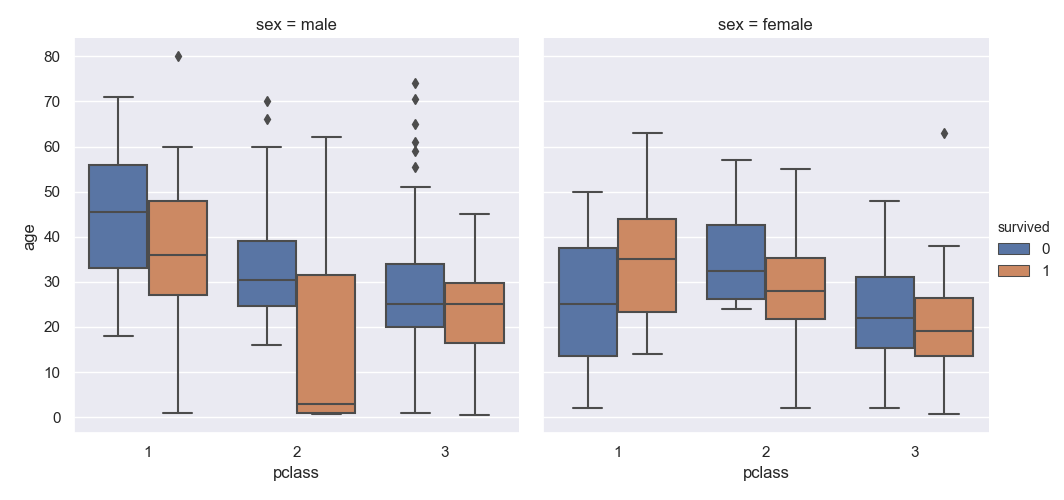
ストリップチャートやスウォームチャートに比べると、ざっくり内容を把握するには良い反面、個々の要素は細かく見ることができない。
violin plot (バイオリン図)
続いては箱ひげ図とスウォームチャートの中間みたいなバイオリン図。
バイオリン図は violinplot() を使って描く。
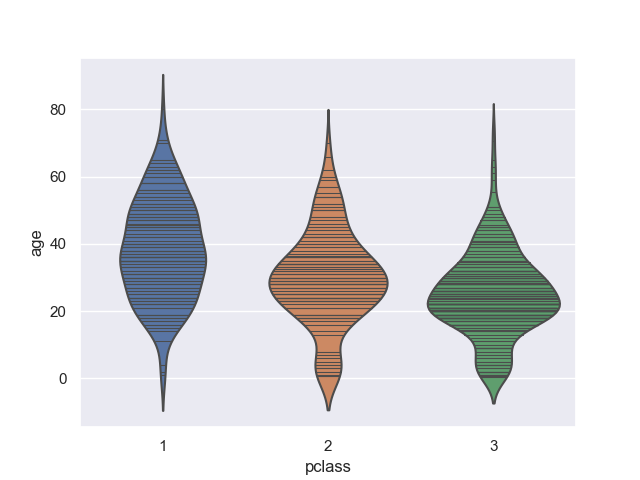
>>> sns.violinplot(data=titanic, x='pclass', y='age')
<matplotlib.axes._subplots.AxesSubplot object at 0x11d70ebe0>
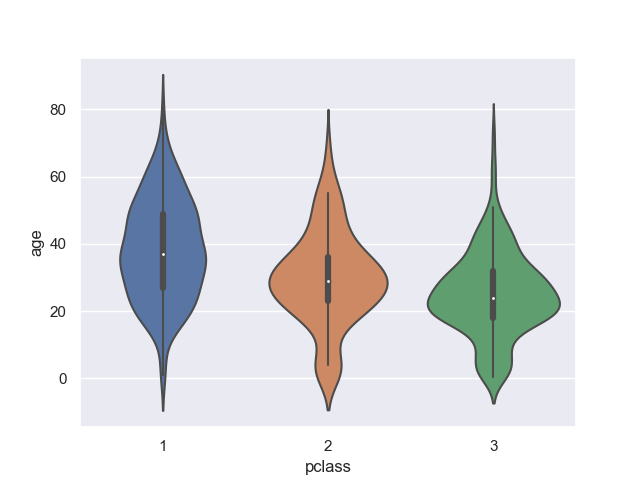
バイオリンの内側については描き方がいくつか考えられる。
例えば inner オプションに stick を指定すると、以下のように個々の要素がどこにあるか示される。
>>> sns.violinplot(data=titanic, x='pclass', y='age', inner='stick')
<matplotlib.axes._subplots.AxesSubplot object at 0x11e572f60>
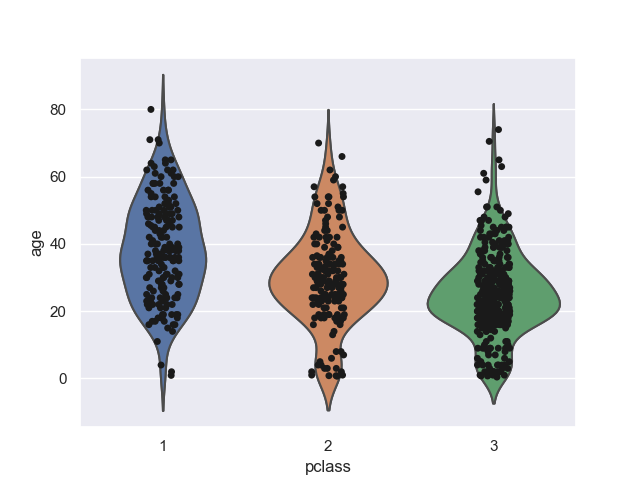
あるいは、次のようにしてグラフを重ね合わせて自分で描いても良い。
>>> ax = sns.violinplot(data=titanic, x='pclass', y='age', inner=None)
>>> sns.stripplot(data=titanic, x='pclass', y='age', color='k', ax=ax)
<matplotlib.axes._subplots.AxesSubplot object at 0x11ec321d0>
層化させたときの表示方法も複数ある。
hue オプション以外、特に何も指定しなければ次のようになる。
箱ひげ図などと同じ感じ。
>>> sns.violinplot(data=titanic, x='pclass', y='age', hue='survived')
<matplotlib.axes._subplots.AxesSubplot object at 0x11d7e6780>
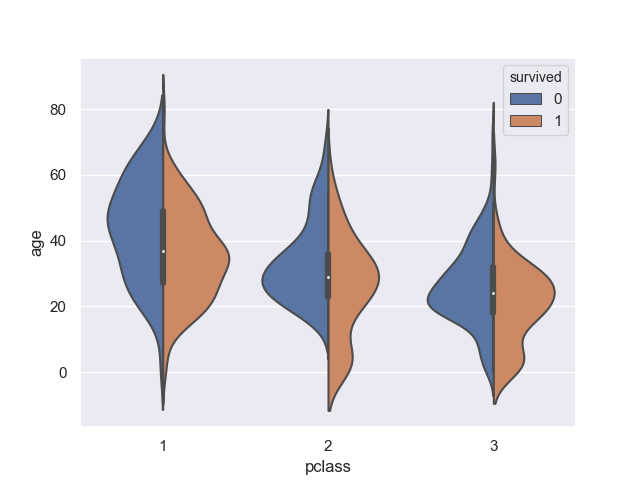
ここで、同時に split オプションに True を指定すると、次のように左右で表示が変わる。
>>> sns.violinplot(data=titanic, x='pclass', y='age', hue='survived', split=True)
<matplotlib.axes._subplots.AxesSubplot object at 0x11e94eb38>
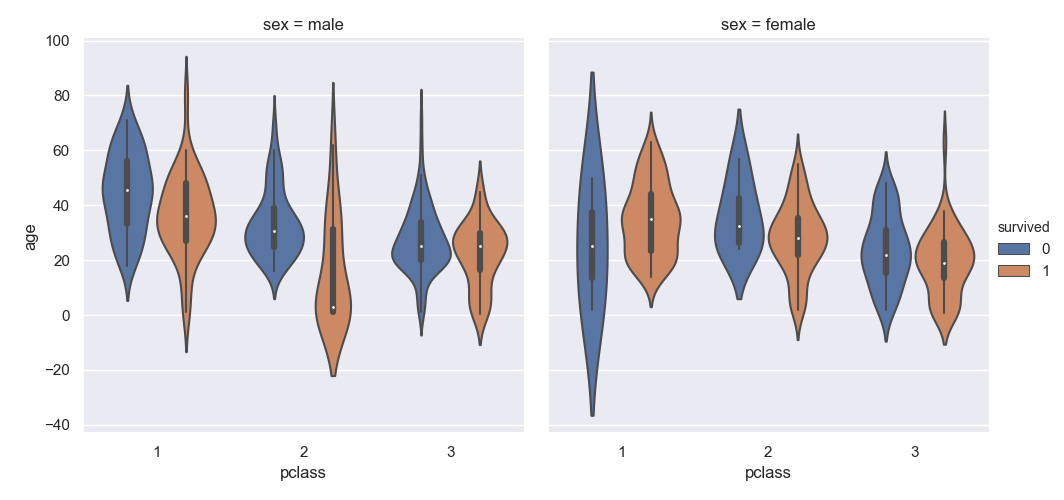
複数のグラフに分けるときは、これまでと同じように catplot() を指定する。
kind オプションには violin を指定する。
>>> sns.catplot(data=titanic, kind='violin', x='pclass', y='age', hue='survived', col='sex')
<seaborn.axisgrid.FacetGrid object at 0x11e127198>
boxen plot (a.k.a letter value plot)
日本語の対応が不明なんだけど、箱ひげ図を改良したグラフ。
一般的には "letter value plot" と呼ばれているみたい。
seaborn では boxenplot() 関数を使って描く。
>>> sns.boxenplot(data=titanic, x='pclass', y='age')
<matplotlib.axes._subplots.AxesSubplot object at 0x10cb33710>
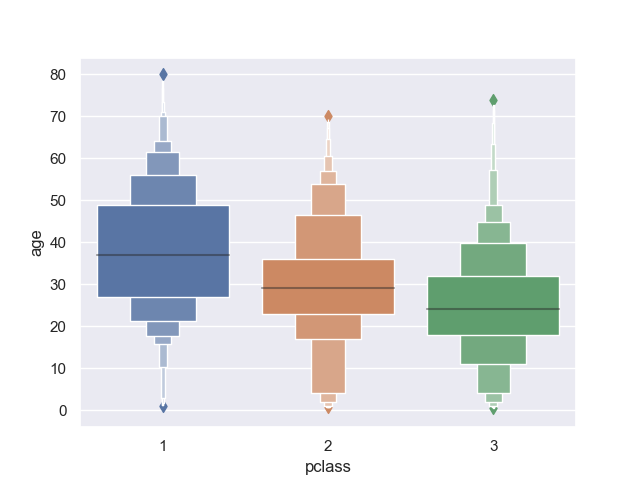
箱ひげ図よりも分布に関する情報の落ち方が少ないのがポイントらしい。
複数のグラフに分けるときは catplot() 関数で kind に boxen を指定する。
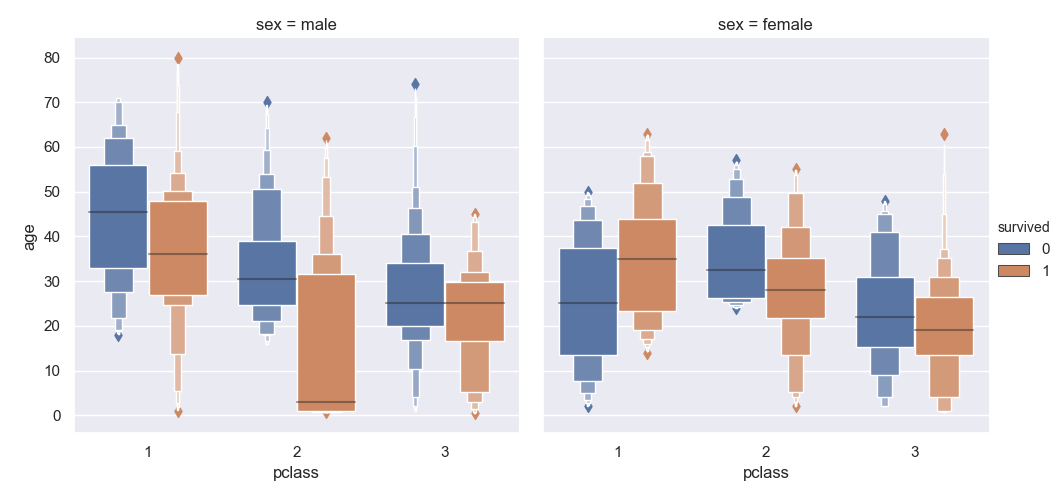
>>> sns.catplot(data=titanic, kind='boxen', x='pclass', y='age', hue='survived', col='sex')
<seaborn.axisgrid.FacetGrid object at 0x11e0a2d68>
point plot
こちらも日本語の対応が分からない。
平均値と信頼区間だけの表示に絞られたシンプルなグラフ。
>>> sns.pointplot(data=titanic, x='pclass', y='age')
<matplotlib.axes._subplots.AxesSubplot object at 0x11d459d30>
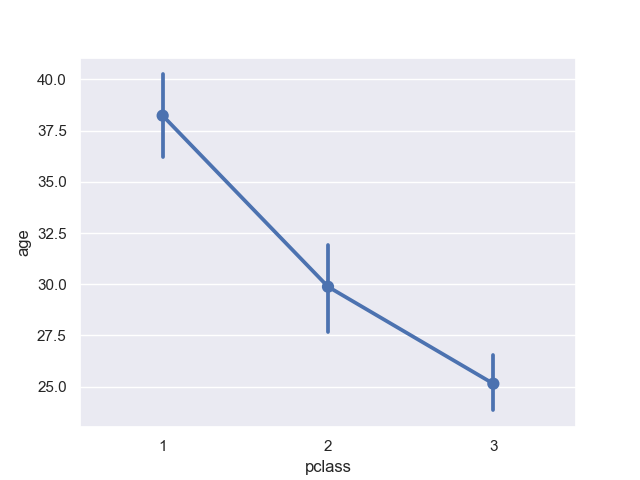
シンプルがゆえに、層化すると統計的に有意か否かを示しやすいかも。
そういえば効果を示すときにこんなグラフ使ってるの見たことあるな。
>>> sns.pointplot(data=titanic, x='pclass', y='age', hue='sex')
<matplotlib.axes._subplots.AxesSubplot object at 0x11d4b2278>
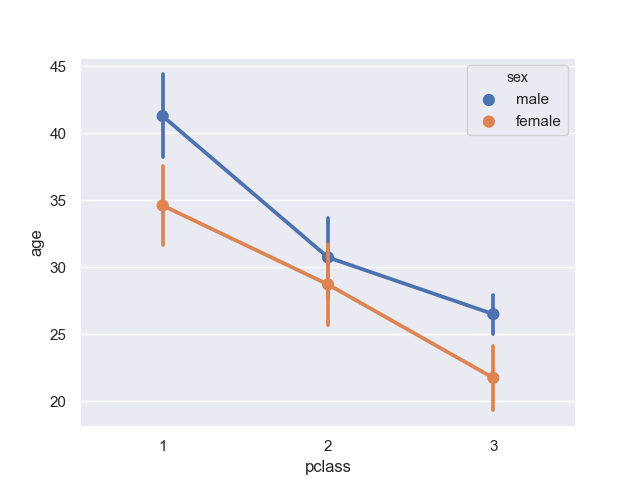
複数のグラフに分けるときは catplot() 関数で kind に point を指定する。
>>> sns.catplot(data=titanic, kind='point', x='pclass', y='age', hue='survived', col='sex')
<seaborn.axisgrid.FacetGrid object at 0x11d456080>
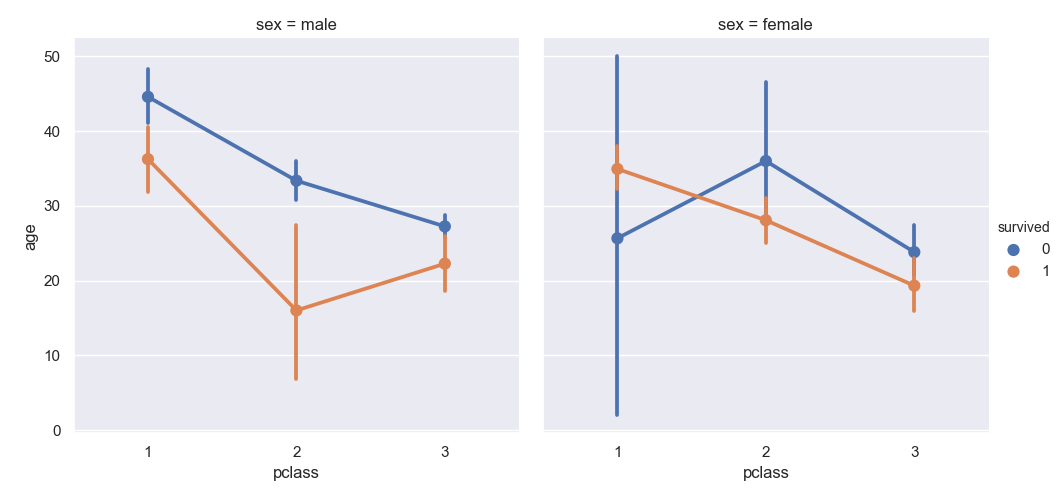
barplot (棒グラフ)
馴染みのある棒グラフ。
>>> sns.barplot(data=titanic, x='pclass', y='age')
<matplotlib.axes._subplots.AxesSubplot object at 0x11e65d080>
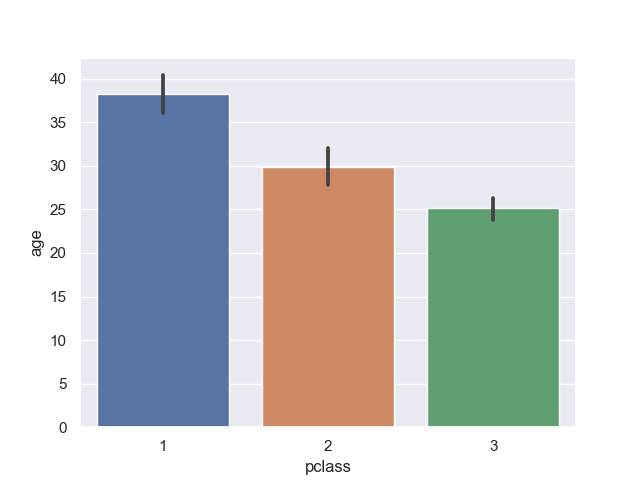
ひげはブートストラップ信頼区間を示している。
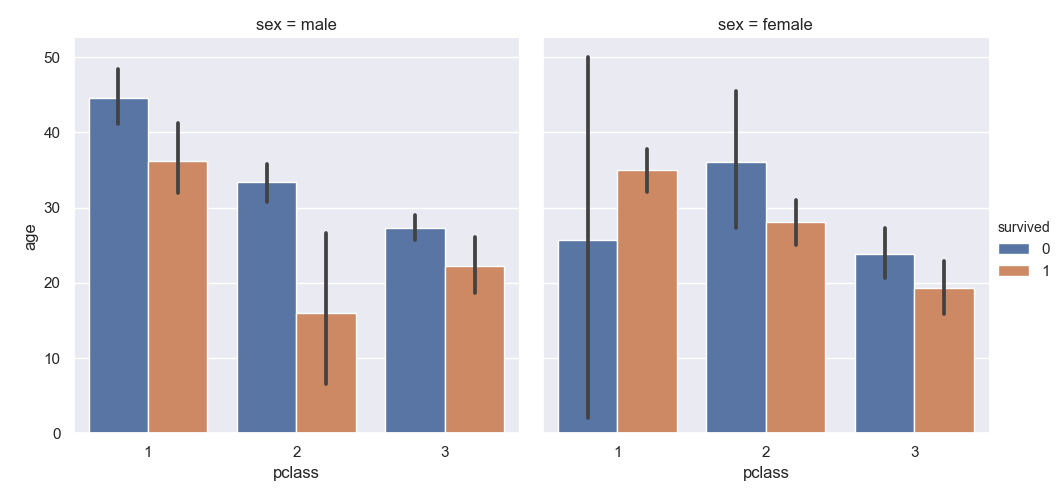
複数のグラフに分けるときは catplot() 関数で kind に bar を指定する。
>>> sns.catplot(data=titanic, kind='bar', x='pclass', y='age', hue='survived', col='sex')
<seaborn.axisgrid.FacetGrid object at 0x11d6eaac8>
count plot
同じ棒グラフでも値のカウントに特価したのが、この countplot() 関数。
使うときは x 軸か y 軸の一軸だけを指定する。
>>> sns.countplot(data=titanic, x='pclass')
<matplotlib.axes._subplots.AxesSubplot object at 0x11da03978>
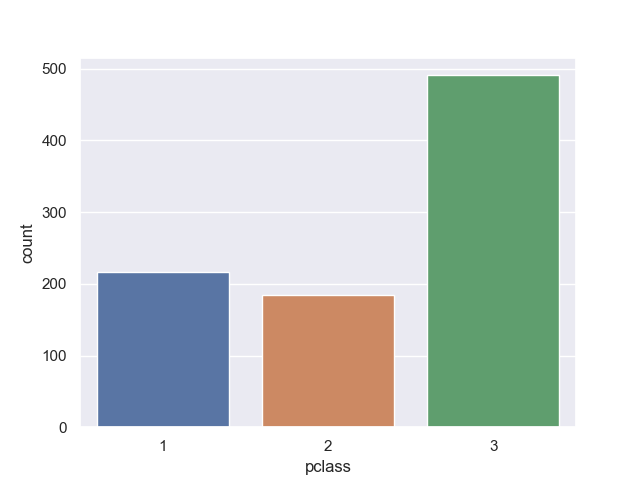
比率などに焦点を絞って可視化するときに見やすい。
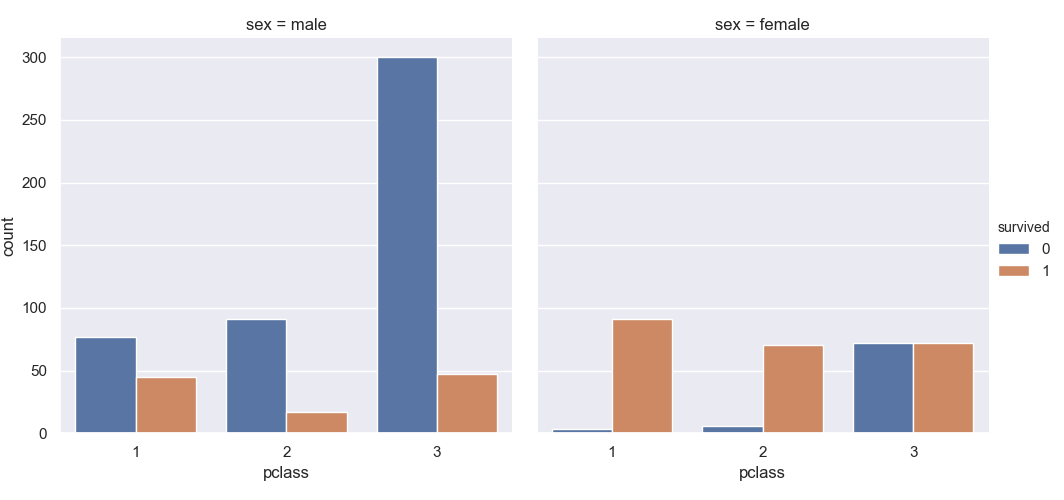
>>> sns.catplot(data=titanic, kind='count', x='pclass', hue='survived', col='sex')
<seaborn.axisgrid.FacetGrid object at 0x11e09a198>
Distribution plots
続いては "Distribution plots" に分類されるグラフを見ていく。
動作確認用として "iris" データセットを読み込んでおく。
>>> iris = sns.load_dataset('iris')
>>> iris.head()
sepal_length sepal_width petal_length petal_width species
0 5.1 3.5 1.4 0.2 setosa
1 4.9 3.0 1.4 0.2 setosa
2 4.7 3.2 1.3 0.2 setosa
3 4.6 3.1 1.5 0.2 setosa
4 5.0 3.6 1.4 0.2 setosa
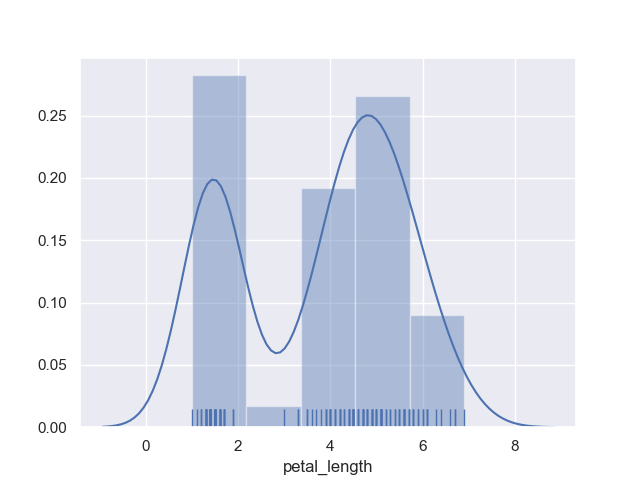
dist plot (ヒストグラム)
まずは馴染みの深いヒストグラムから。
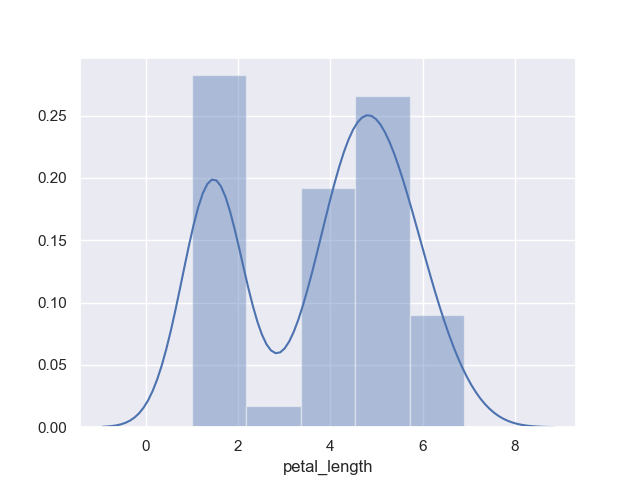
ヒストグラムは distplot() 関数を使って描画する。
>>> sns.distplot(iris.petal_length)
<matplotlib.axes._subplots.AxesSubplot object at 0x11ee27160>
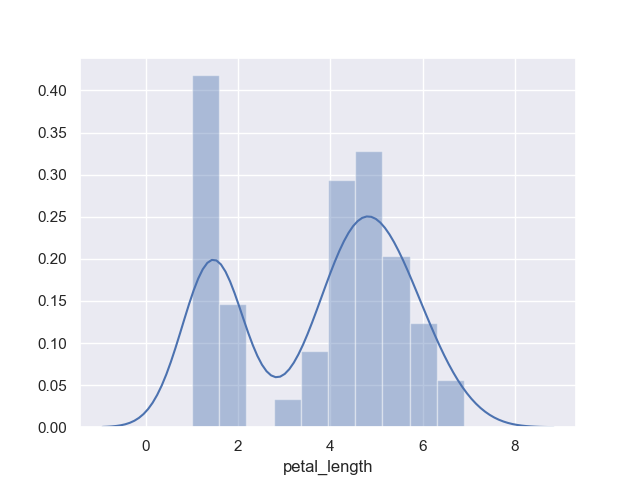
階級の数は bins オプションで指定できる。
>>> sns.distplot(iris.petal_length, bins=10)
<matplotlib.axes._subplots.AxesSubplot object at 0x11e0ced68>
kde plot
KDE (Kernel Density Estimation) はカーネル密度推定という。
分布から確率密度関数を推定するのに用いる。
>>> sns.kdeplot(iris.sepal_length)
<matplotlib.axes._subplots.AxesSubplot object at 0x11d34e160>
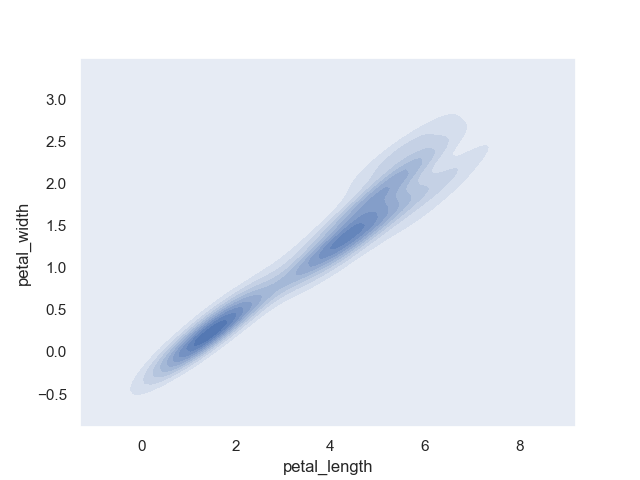
二軸で描画することもできる。
>>> sns.kdeplot(iris.petal_length, iris.petal_width, shade=True)
<matplotlib.axes._subplots.AxesSubplot object at 0x11c5832b0>
rug plot
rug plot は値の登場する位置に特化したグラフ。
>>> sns.rugplot(iris.petal_length)
<matplotlib.axes._subplots.AxesSubplot object at 0x11c701ba8>
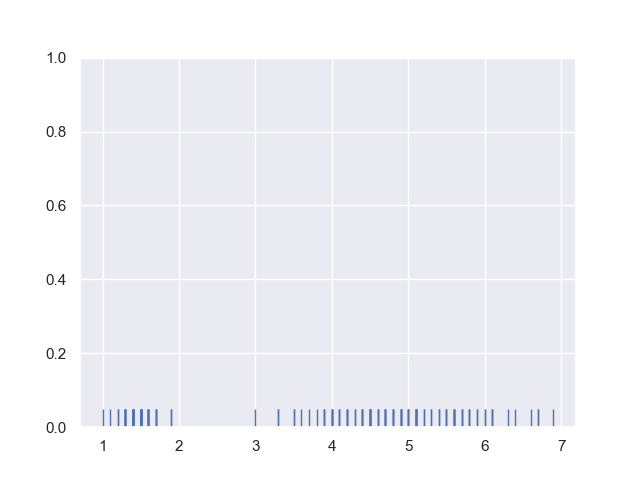
どちらかというと、他のグラフと重ね合わせて使うものなのかな。
>>> ax = sns.distplot(iris.petal_length)
>>> sns.rugplot(iris.petal_length, ax=ax)
<matplotlib.axes._subplots.AxesSubplot object at 0x11e323c88>
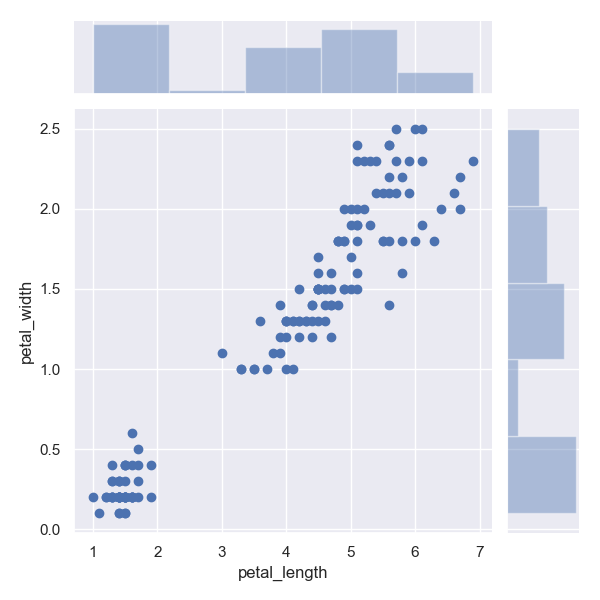
joint plot
joint plot は二つのグラフの組み合わせ。
デフォルトでは散布図とヒストグラムが同時に見られる。
>>> sns.jointplot(data=iris, x='petal_length', y='petal_width')
<seaborn.axisgrid.JointGrid object at 0x11c6d8320>
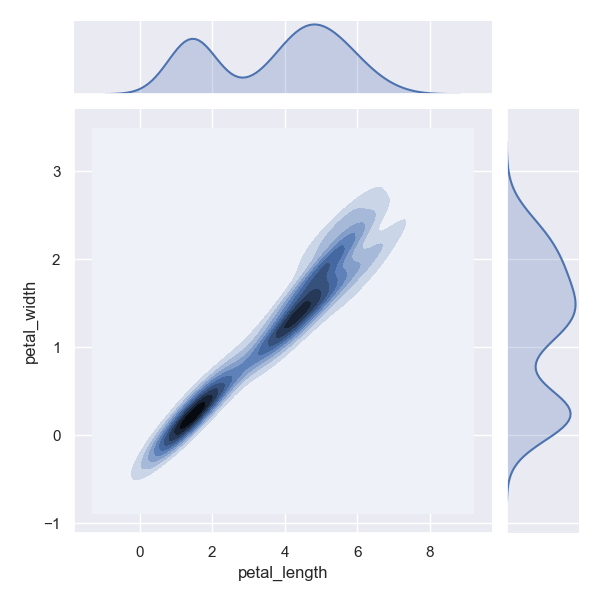
kind に kde を指定すると確率密度関数が見られる。
>>> sns.jointplot(data=iris, x='petal_length', y='petal_width', kind='kde')
<seaborn.axisgrid.JointGrid object at 0x11e6635c0>
pair plot
pair plot は二軸の組み合わせについて可視化できる。
>>> sns.pairplot(data=iris)
<seaborn.axisgrid.PairGrid object at 0x11e6d6470>
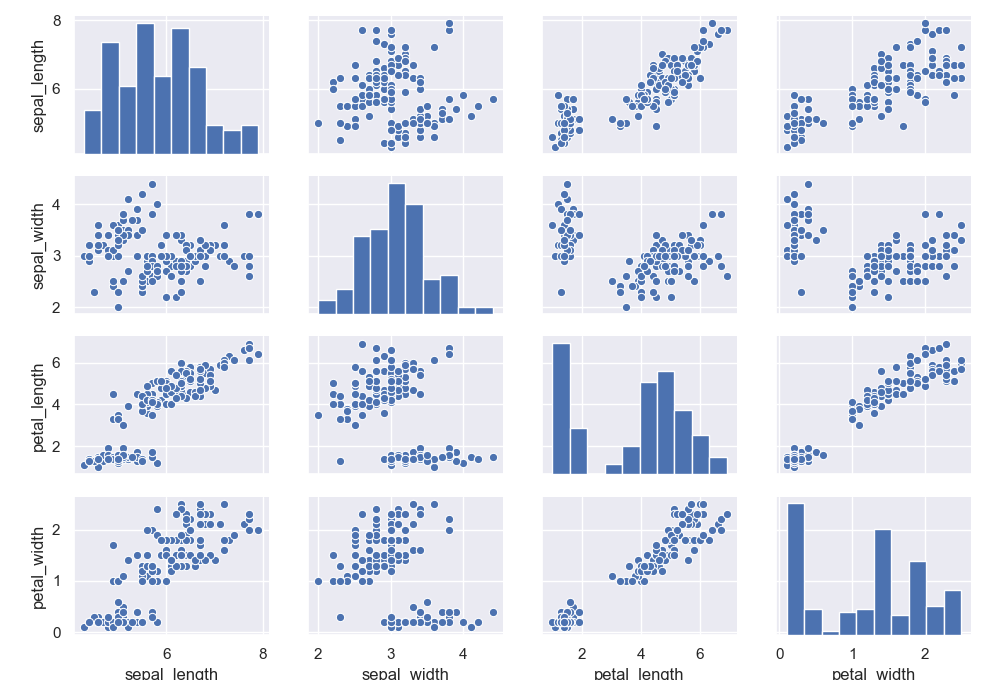
表示する次元を絞るときは vars オプションで指定する。
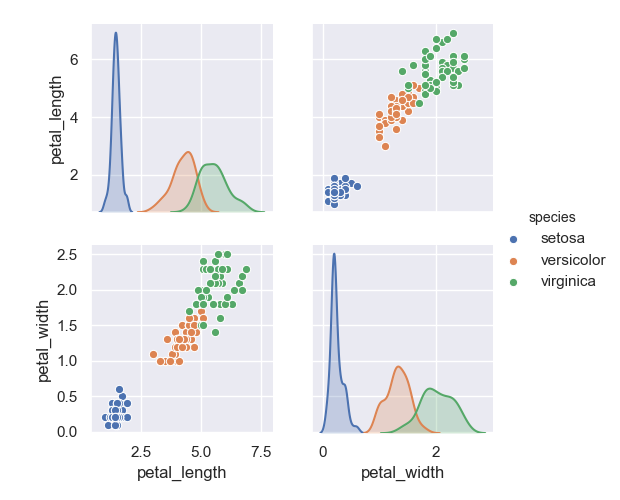
>>> sns.pairplot(data=iris, hue='species', vars=['petal_length', 'petal_width'])
<seaborn.axisgrid.PairGrid object at 0x11e565390>
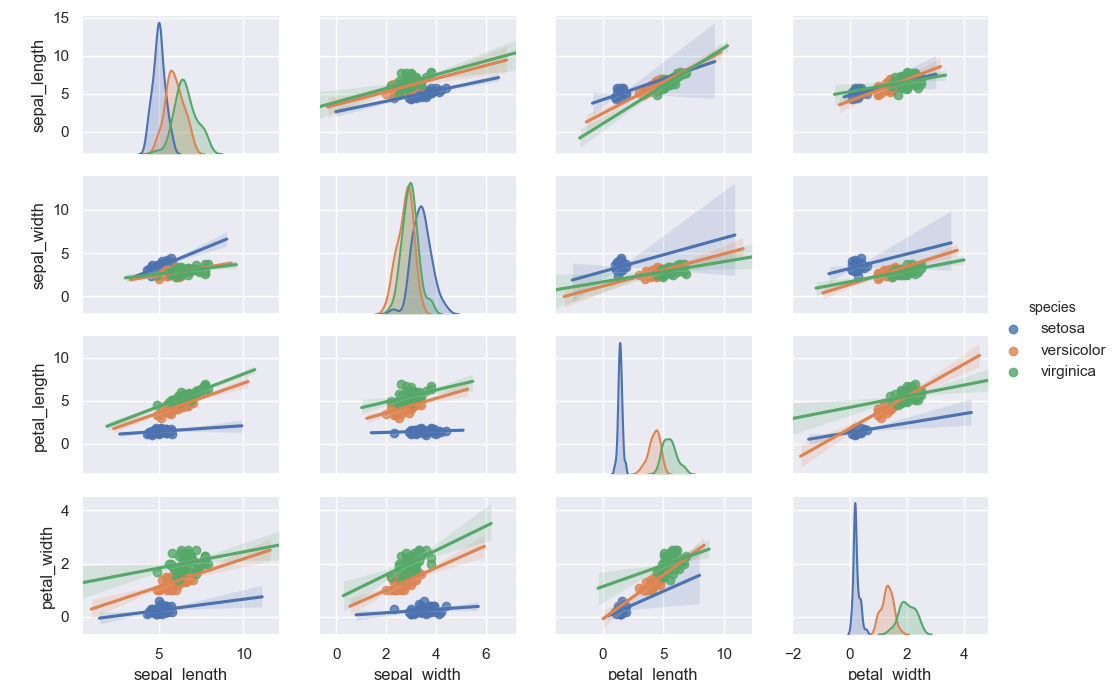
kind オプションに reg を指定すると線形回帰の結果も見られたりする。
>>> sns.pairplot(data=iris, hue='species', kind='reg')
<seaborn.axisgrid.PairGrid object at 0x11db1a668>
Matrix plots
続いては "Matrix plots" に分類されるグラフを見ていく。
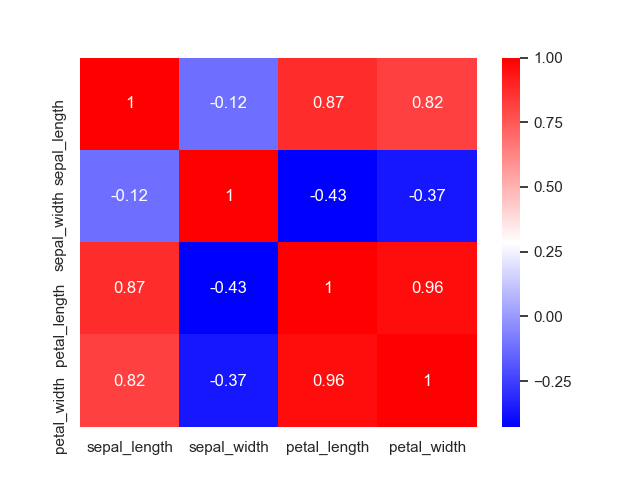
heat map (ヒートマップ)
まずはヒートマップから。
相関係数を確認するのに使うことが多いと思う。
>>> sns.heatmap(data=iris.corr())
<matplotlib.axes._subplots.AxesSubplot object at 0x11d8d2048>
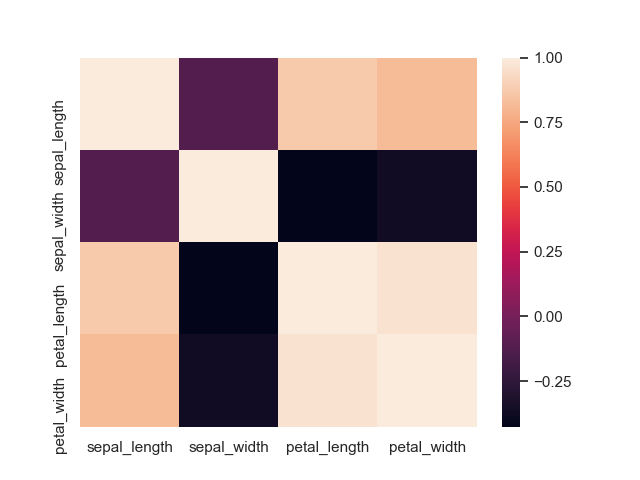
実際の値も一緒に描いたり、カラーマップを変更すると見やすくなる。
>>> sns.heatmap(data=iris.corr(), annot=True, cmap='bwr')
<matplotlib.axes._subplots.AxesSubplot object at 0x11da5cac8>
まとめ
今回は searborn を使って色々なグラフを描いてみた。
seaborn は多くの API が共通のオプションを備えているため、それらを覚えるだけでなんとなく描けるようになるところが便利。