今回は、ハイパーパラメータ選びを含む機械学習モデルの交差検証について書いてみる。 このとき、交差検証のやり方がまずいと汎化性能を本来よりも高く見積もってしまう恐れがある。 汎化性能というのは、未知のデータに対処する能力のことを指す。 ようするに、いざモデルを実環境に投入してみたら想定よりも性能が出ない (Underperform) ということが起こる。 これを防ぐには、交差検証の中でも Nested Cross Validation (Nested CV) あるいは Double Cross Validation と呼ばれる手法を使う。
ハイパーパラメータの選び方としては、色々な組み合わせをとにかく試すグリッドサーチという方法を例にする。 また、モデルのアルゴリズムにはサポートベクターマシンを使った。 これは、サポートベクターマシンはハイパーパラメータの変更に対して敏感な印象があるため。
その他、使った環境は次の通り。
$ sw_vers ProductName: Mac OS X ProductVersion: 10.13.5 BuildVersion: 17F77 $ python -V Python 3.6.5
下準備
まずは、今回のサンプルコードで使うパッケージをあらかじめインストールしておく。
$ pip install scikit-learn numpy scipy tqdm matplotlib
続いて Python の REPL を起動する。
$ python
起動したら scikit-learn に組み込みで用意されている乳がんデータセットを読み込んでおこう。 これには、しこりに関する特徴量とそれが良性か悪性かの情報が含まれる。
>>> from sklearn import datasets >>> >>> dataset = datasets.load_breast_cancer() >>> X = dataset.data >>> y = dataset.target
これで準備が整った。
ここからは、前提となる知識として交差検証に至る機械学習モデルを評価するやり方を一つずつ紹介していく。 おそらく、知っている内容も多いと思うので必要に応じて読み飛ばしてもらえると。
学習に用いたデータでモデルを評価する
まずは、最もダメなパターンから。 これは、モデルの学習に用いたデータを使って、そのモデルを評価するというもの。 これをやってしまうと、汎化性能は全く測れない。 なにせ全然未知ではなく、モデルが既に見たことのあるデータなのだから。
概念図はこんな感じ。

とはいえ、ダメなパターンについても見ておくことは重要なので以下にサンプルコードを示す。 まずはサポートベクターマシンの分類器を用意して、データセットを全て使って学習させる。
>>> from sklearn.svm import SVC >>> >>> svm = SVC(kernel='rbf') >>> svm.fit(X, y) SVC(C=1.0, cache_size=200, class_weight=None, coef0=0.0, decision_function_shape='ovr', degree=3, gamma='auto', kernel='rbf', max_iter=-1, probability=False, random_state=None, shrinking=True, tol=0.001, verbose=False)
続いて、学習したモデルを使ってデータセット全てに対して予測する。
>>> y_pred = svm.predict(X)
これは、学習に使ったデータを、そのまま予測しているということ。
予測結果の精度 (Accuracy) を計算してみよう。
>>> from sklearn.metrics import accuracy_score >>> >>> accuracy_score(y, y_pred) 1.0
なんと 100% の精度が得られた!ヤッター! …といっても、これはモデルが一度見たことのあるデータを予測しているだけなので、何ら驚くには値しない。 もちろん、これではモデルの汎化性能は測れない。
ホールドアウト検証 (Hold-out Validation)
続いては、汎化性能をそれなりに評価するための方法としてホールドアウト検証を紹介する。 これは、データセットを学習用とテスト用に分割する。 そして、学習用のデータをモデルに学習させた上で、テスト用のデータを使ってモデルの性能を評価するというもの。 テスト用のデータはモデルにとって見たことのない未知のデータなので、これは汎化性能を示す指標となりうる。
概念図はこんな感じ。

先ほどと同様に、サンプルコードを示す。 まずは、データセットを学習用とテスト用に分割する。
>>> from sklearn.model_selection import train_test_split >>> >>> X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, shuffle=True, random_state=42)
分割方法に再現性を持たせたい場合には random_state オプションを指定した方が良い。
この数値を指定して、他のオプションについても値が同じである限りデータが同じように分割される。
また、データの分割に偏りを作らないためには shuffle オプションを有効にしてランダムにデータを選択した方が良い。
もちろん、ただランダムに分割するだけでは偏りを取り除けない場合には、それ以外のやり方で分割する必要がある。
データを分割したら、学習用データの方を使ってモデルを学習する。
>>> svm = SVC(kernel='rbf') >>> svm.fit(X_train, y_train) SVC(C=1.0, cache_size=200, class_weight=None, coef0=0.0, decision_function_shape='ovr', degree=3, gamma='auto', kernel='rbf', max_iter=-1, probability=False, random_state=None, shrinking=True, tol=0.001, verbose=False)
今度は 64.3% の精度が得られた。 こちらの方が、先ほどよりもモデルの性能を現実的に示している。
>>> y_pred = svm.predict(X_test)
>>> accuracy_score(y_test, y_pred)
0.6436170212765957
交差検証 (Cross Validation)
ホールドアウト検証法はデータを分割しているとはいっても一回だけの試行なので偏りが含まれる余地が比較的ある。 この偏りを減らすには交差検証というやり方を用いる。 これは、複数回に渡って異なる分割をしたデータに対し、それぞれでホールドアウト検証をして結果を合算するというもの。
概念図はこんな感じ。

scikit-learn では KFold を使うと交差検証が楽にできる。
以下のサンプルコードでは分割数 (試行回数) として 4 を指定した。
>>> from sklearn.model_selection import KFold >>> >>> kf = KFold(n_splits=4, shuffle=True, random_state=42)
交差検証のスコアは cross_val_score を使うと楽に計算できる。
といっても、これは先ほどのホールドアウト検証を KFold を使ってループしながら実行しているだけ。
自分で書いても全然問題はない。
>>> from sklearn.model_selection import cross_val_score >>> >>> svm = SVC(kernel='rbf') >>> scores = cross_val_score(svm, X=X, y=y, cv=kf)
結果としては、各ホールドアウト検証における性能が得られる。
>>> scores array([0.62237762, 0.69014085, 0.61971831, 0.57746479])
一般的には、上記を単純に算術平均すると思う。
>>> average_score = scores.mean()
>>> average_score
0.6274253915098986
ちなみに分割数をデータ点数まで増やした場合は Leave-One-Out 検証法と呼ばれる。 機械学習系の文章の中では、よく LOO と省略されていることがある。
ハイパーパラメータの選択を含む交差検証
ここからが本題。 先ほどのサンプルコードでは、基本的にサポートベクターマシンのモデルをデフォルトのハイパーパラメータで扱っていた。 ただ、実際に使うときはハイパーパラメータの調整が必要になる。 このとき、ただ単純に交差検証をするだけだとモデルの性能を高く見積もってしまう恐れがある。
上記をサンプルコードと共に確認する。 まずは、先ほどと同じようにサポートベクターマシンのモデルと交差検証用のオブジェクトを用意する。
>>> svm = SVC(kernel='rbf') >>> kf = KFold(n_splits=4, shuffle=True, random_state=42)
ハイパーパラメータの候補は、次のように辞書とリストを組み合わせて用意する。
>>> candidate_params = {
... 'C': [1, 10, 100],
... 'gamma': [0.01, 0.1, 1],
... }
GridSearchCV にモデルとハイパーパラメータの候補を渡して、データを学習させる。
GridSearchCV は名前に CV と入っている通り、内部的に交差検証を使いながら性能の良いハイパーパラメータの組み合わせを探してくれる。
>>> from sklearn.model_selection import GridSearchCV >>> from multiprocessing import cpu_count >>> >>> gs = GridSearchCV(estimator=svm, param_grid=candidate_params, cv=kf, n_jobs=cpu_count()) >>> gs.fit(X, y) GridSearchCV(cv=KFold(n_splits=4, random_state=42, shuffle=True), error_score='raise', estimator=SVC(C=1.0, cache_size=200, class_weight=None, coef0=0.0, decision_function_shape='ovr', degree=3, gamma='auto', kernel='rbf', max_iter=-1, probability=False, random_state=None, shrinking=True, tol=0.001, verbose=False), fit_params=None, iid=True, n_jobs=4, param_grid={'C': [1, 10, 100], 'gamma': [0.01, 0.1, 1]}, pre_dispatch='2*n_jobs', refit=True, return_train_score='warn', scoring=None, verbose=0)
学習が終わると、最も性能の良かったハイパーパラメータで学習したモデルが GridSearchCV#best_estimator_ で得られる。
>>> gs.best_estimator_ SVC(C=10, cache_size=200, class_weight=None, coef0=0.0, decision_function_shape='ovr', degree=3, gamma=0.01, kernel='rbf', max_iter=-1, probability=False, random_state=None, shrinking=True, tol=0.001, verbose=False)
同様に GridSearchCV#best_params_ で最も性能の良かったハイパーパラメータの組み合わせが得られる。
>>> gs.best_params_
{'C': 10, 'gamma': 0.01}
また、GridSearchCV#best_score_ から上記のモデルが記録したスコアも得られる。
>>> gs.best_score_
0.6344463971880492
では、上記のハイパーパラメータを使ったモデルなら未知のデータに対して 63.4% の精度が得られるはずかというと、そうでもないらしい。 これは、一回の交差検証だけだと精度が偏って得られることも考えられるため。 ようするに、大きく外してはいないはずだけど見積もりとしては楽観的なものになる。 この、一回の交差検証だけで評価するやり方を Non-nested Cross Validation (Non-nested CV) という。
概念図としてはこんな感じ。 さっきの単純な交差検証をハイパーパラメータの組み合わせごとにやっているだけ。
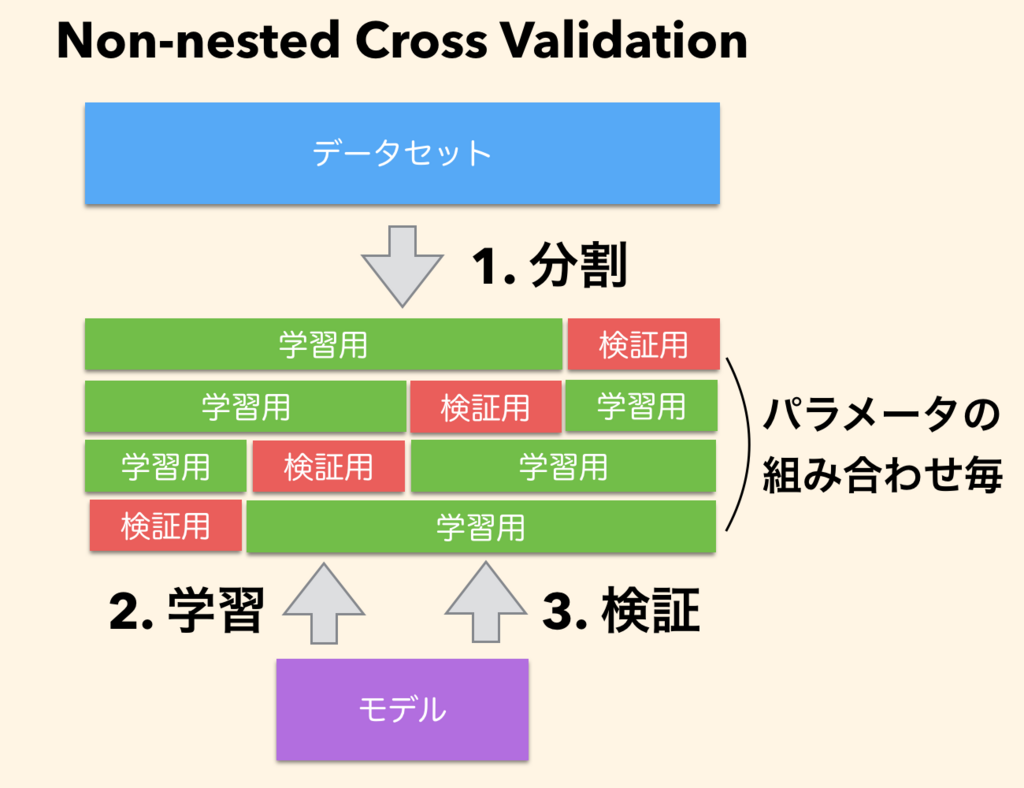
Nested Cross Validation (Nested CV)
前述した問題をどうやって解決するかというと、交差検証を二重にする。 この方法は Nested CV と呼ばれる。
概念図はこんな感じ。

Nested CV では、交差検証を内側 (Inner CV) と外側 (Outer CV) の二重に分けている。 内側ではハイパーパラメータの選択に注力し、外側はできたモデルの評価に注力する。 ポイントとしては、それぞれで重複するデータをモデルに触らせていないところ。 一度でもモデルに見せたデータはその時点で汚れてしまうため、評価する上で二度と使うことはできない。
サンプルコードで Nested CV を見ていこう。 まずは、先ほどと同じようにグリッドサーチ用のオブジェクトまで作っておく。
>>> svm = SVC(kernel='rbf') >>> kf = KFold(n_splits=4, shuffle=True, random_state=42) >>> gs = GridSearchCV(estimator=svm, param_grid=candidate_params, cv=kf, n_jobs=cpu_count())
続いて、上記の GridSearchCV のインスタンスをさらに cross_val_score() 関数に突っ込む。
>>> scores = cross_val_score(gs, X=X, y=y, cv=kf)
上記は正直なかなか分かりにくいので、順を追って解説する。
まず、cross_val_score() 関数が前述した外側の交差検証になっている。
外側で分割した学習用データのみが GridSearchCV のインスタンスに渡される。
GridSearchCV のインスタンスは、渡された学習用データをさらに分割して学習用データとハイパーパラメータ調整用データにする。
そして、そのデータを使ってハイパーパラメータを選択する。
これが前述した内側の交差検証になる。
ハイパーパラメータの選択が終わったら、できあがったモデルが外側の交差検証で評価される。
ようするに、外側の交差検証の分割数×内側の交差検証の分割数×ハイパーパラメータの組み合わせの数だけホールドアウト検証を繰り返すことになる。
上記で得られたスコアが以下の通り。 ようするに、これは各内側の交差検証で性能の良かったモデルたちが外側の交差検証で記録した性能ということになる。
>>> scores array([0.62937063, 0.69014085, 0.61971831, 0.58450704])
上記の算術平均は次の通り。 先ほどの Non-nested CV よりも、ほんの少しではあるが下がっている。
>>> average_score = scores.mean()
>>> average_score
0.6309342066384319
Non-nested CV に比べると、この Nested CV で記録した値の方が現実に則した汎化性能を表している、とされる。
Non-nested CV と Nested CV が記録するスコアを比較する
先ほどの例では Non-nested CV よりも Nested CV の方が低めのスコアが出た。 一回だけならたまたまということも考えられるので、念のため何度か試行してグラフにプロットしてみる。
次のサンプルコードでは Non-nested CV と Nested CV を 50 回繰り返して、それぞれが記録するスコアをプロットする。
#!/usr/bin/env python # -*- coding: utf-8 -*- from multiprocessing import cpu_count from sklearn import datasets from sklearn.svm import SVC from sklearn.model_selection import KFold from sklearn.model_selection import cross_val_score from sklearn.model_selection import GridSearchCV import numpy as np from matplotlib import pyplot as plt from tqdm import tqdm def main(): NUM_TRIALS = 50 # データセットを読み込む dataset = datasets.load_breast_cancer() X = dataset.data y = dataset.target # 候補となるハイパーパラメータ candidate_params = { 'C': [1, 10, 100], 'gamma': [0.01, 0.1, 1, 'auto'], } # 計測したスコアを保存するためのリスト scores_non_nested_cv = np.zeros(NUM_TRIALS) scores_nested_cv = np.zeros(NUM_TRIALS) # 何回か試してみる for i in tqdm(range(NUM_TRIALS)): # Non Nested CV svm = SVC(kernel='rbf') kf = KFold(n_splits=4, shuffle=True, random_state=i) gscv = GridSearchCV(estimator=svm, param_grid=candidate_params, cv=kf, n_jobs=cpu_count()) gscv.fit(X, y) scores_non_nested_cv[i] = gscv.best_score_ # Nested CV svm = SVC(kernel='rbf') kf = KFold(n_splits=4, shuffle=True, random_state=i) gs = GridSearchCV(estimator=svm, param_grid=candidate_params, cv=kf, n_jobs=cpu_count()) scores = cross_val_score(gs, X=X, y=y, cv=kf) scores_nested_cv[i] = scores.mean() # スコア平均と標準偏差 print('non nested cv: mean={:.5f} std={:.5f}'.format(scores_non_nested_cv.mean(), scores_non_nested_cv.std())) print('nested cv: mean={:.5f} std={:.5f}'.format(scores_nested_cv.mean(), scores_nested_cv.std())) # グラフを描画する plt.figure(figsize=(10, 6)) plt.plot(scores_non_nested_cv, color='g', label='non nested cv') plt.plot(scores_nested_cv, color='b', label='nested cv') plt.legend() plt.show() if __name__ == '__main__': main()
適当な名前をつけて上記を保存したら実行してみよう。
$ python cv.py 36%|█████████████████▎ | 18/50 [00:47<01:24, 2.64s/it]
計算には時間が結構かかるので tqdm を使って進捗を表示させている。
tqdm については、以前に以下の記事で紹介している。
50 回試した上での精度の平均は次の通り。 Nested CV の方が 0.3% ほど精度を低く見積もっていることが分かる。
non nested cv: mean=0.63195 std=0.00261 nested cv: mean=0.62878 std=0.00255
得られたグラフは次の通り。 一部に例外はあるものの、基本的には Nested CV の方が Non-nested CV よりも精度を低く見積もっている。

疑問と悩み
めでたしめでたし。 と、言いたいところなんだけど、いくつか自分でもまだ完全には腑に落ちていないところがある。
学習に使うデータが減る問題
Nested CV の方が Non-nested CV よりも精度の見積もりは低く出た。 とはいえ Nested CV では Non-nested CV よりもモデルの学習に使うデータ自体も減っている。 これは、データの分割が Non-nested CV では二つなのに対して Nested CV では三つになっているため。 具体的には Nested CV ではデータを学習用、ハイパーパラメータ調整用、検証用に分割することになる。 対して Non-nested CV では学習用と検証用にしか分割していない。
学習に使うデータが減れば、バイアス以外にもそれだけで精度が低くなる余地があるように感じる。 かといって、ハイパーパラメータ調整用や検証用のデータを減らすと、精度の分散が大きくなってモデル選択が難しくなるような気がする。 まあ、もちろんそれで Nested CV をやらない理由にはならないだろうけど。
一体どのハイパーパラメータを選べば良いのよ問題
Nested CV では、内側の交差検証で選ばれてくるモデルたちのハイパーパラメータがどれも同一とは限らないはず。 同一でない場合には、結局のところどのハイパーパラメータの組み合わせを選べば良いのよ?となる。 まあ、本当にバラバラならどれを使っても似たようなものなんだ、という理解にはつながるかもしれないけど。
もし結果をそのまま使いたいなら、内側の交差検証で選ばれた各モデルを使ってアンサンブル (Voting) すると良いのかな? 実際のところ、ハイパーパラメータの目星がついたからといって、改めてモデルに未分割の全データを学習させて同じ汎化性能が得られるとは限らない。 交差検証をしていないモデルからは、どんな結果が得られてもおかしくはないのだから。
参考
Nested versus non-nested cross-validation — scikit-learn 0.19.2 documentation
Nested Cross Validation: When Cross Validation Isn’t Enough
いじょう。

スマートPythonプログラミング: Pythonのより良い書き方を学ぶ
- 作者: もみじあめ
- 発売日: 2016/03/12
- メディア: Kindle版
- この商品を含むブログ (1件) を見る