今回は RFE (Recursive Feature Elimination) と呼ばれる手法を使って特徴量選択 (Feature Selection) してみる。
教師データの中には、モデルの性能に寄与しない特徴量が含まれている場合がある。
アルゴリズムがノイズに対して理想的にロバストであれば、有効な特徴量だけを読み取って学習するため特徴量選択は不要かもしれない。
しかしながら、現実的にはそのような仮定を置くことが難しい場合があると思う。
そこで、元の特徴量からモデルの性能に寄与する部分集合を取り出す作業を特徴量選択という。
特徴量選択の手法には、以下の 3 つがあるようだ。
- フィルター法 (Filter Method)
- ラッパー法 (Wrapper Method)
- 組み込み法 (Embedding Method)
RFE は、上記でいうとラッパー法に分類される。
実際に機械学習のモデルを用いて、部分集合を学習・評価した際に性能が上がるか下がるか確認していく。
フィルター法や組み込み法に比べると必要な計算量は多い。
使った環境は次の通り。
$ sw_vers
ProductName: Mac OS X
ProductVersion: 10.14.4
BuildVersion: 18E226
$ python -V
Python 3.7.3
下準備
まずは scikit-learn と matplotlib をインストールしておく。
$ pip install scikit-learn matplotlib
無意味な特徴量が減ることで性能は上がるのか
特徴量を実際に選択してみる前に、そもそも無意味な特徴量を減らすことでモデルの性能は上がるのか確かめておく。
以下のサンプルコードでは二値分類問題のダミーデータを用意している。
データは 100 次元の特徴量を持つものの、実際に意味を持ったものは 5 次元しか含まれていない。
そのデータを使ってランダムフォレストを学習させて 5-Fold CV で AUC (Area Under the Curve) について評価している。
100 次元全てを使った場合と 5 次元だけ使った場合で、どのような差が出るだろうか。
from sklearn.datasets import make_classification
from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import roc_auc_score
from sklearn.model_selection import cross_val_predict
from sklearn.model_selection import StratifiedKFold
def main():
args = {
'n_samples': 1000,
'n_features': 100,
'n_informative': 5,
'n_redundant': 0,
'n_repeated': 0,
'n_classes': 2,
'random_state': 42,
'shuffle': False,
}
X, y = make_classification(**args)
clf = RandomForestClassifier(n_estimators=100,
random_state=42)
skf = StratifiedKFold(n_splits=5,
shuffle=True,
random_state=42)
y_pred = cross_val_predict(clf, X, y,
cv=skf,
method='predict_proba')
metric = roc_auc_score(y, y_pred[:, 1])
print('All used AUC:', metric)
y_pred = cross_val_predict(clf, X[:, :5], y,
cv=skf,
method='predict_proba')
metric = roc_auc_score(y, y_pred[:, 1])
print('Ideal AUC:', metric)
if __name__ == '__main__':
main()
上記の実行結果は次の通り。
全ての特徴量を使った場合の AUC が 0.947 なのに対して、意味を持った特徴量だけを使った場合には 0.981 というスコアが出ている。
あきらかに、意味を持った特徴量だけを使った方が性能が上がっている。
$ python features.py
All used AUC: 0.9475844521611112
Ideal AUC: 0.9811272823286553
特徴量の重要度を確認する
念のため、ランダムフォレストが特徴量の重要度 (Feature Importance) をどのように認識したかを確認しておこう。
以下のサンプルコードでは、全てのデータを使って学習した際の特徴量の重要度を TOP20 で可視化している。
ちなみに、意味のある特徴量は先頭の 5 次元に固まっている。
import numpy as np
from sklearn.datasets import make_classification
from sklearn.ensemble import RandomForestClassifier
from matplotlib import pyplot as plt
def main():
args = {
'n_samples': 1000,
'n_features': 100,
'n_informative': 5,
'n_redundant': 0,
'n_repeated': 0,
'n_classes': 2,
'random_state': 42,
'shuffle': False,
}
X, y = make_classification(**args)
clf = RandomForestClassifier(n_estimators=100,
random_state=42)
clf.fit(X, y)
importances = clf.feature_importances_
importance_std = np.std([tree.feature_importances_
for tree in clf.estimators_],
axis=0)
indices = np.argsort(importances)[::-1]
rank_n = min(X.shape[1], 20)
print('Feature importance ranking (TOP {rank_n})'.format(rank_n=rank_n))
for i in range(rank_n):
params = {
'rank': i + 1,
'idx': indices[i],
'importance': importances[indices[i]]
}
print('{rank}. feature {idx:02d}: {importance}'.format(**params))
plt.figure(figsize=(8, 32))
plt.barh(range(rank_n),
importances[indices][:rank_n],
color='g',
ecolor='r',
yerr=importance_std[indices][:rank_n],
align='center')
plt.yticks(range(rank_n), indices[:rank_n])
plt.xlabel('Features')
plt.ylabel('Importance')
plt.grid()
plt.show()
if __name__ == '__main__':
main()
上記の実行結果は次の通り。
重要度の上位には、ちゃんと意味もある 5 次元 (00 ~ 04) が選ばれている。
ただ、重要度の桁は違うものの、その他の特徴量にもある程度の重要性を見出してしまっているようだ。
$ python importances.py
Feature importance ranking (TOP 20)
1. feature 00: 0.137486590660634
2. feature 01: 0.08702192608775006
3. feature 03: 0.07472282910658508
4. feature 04: 0.07038842117095266
5. feature 02: 0.02259020059710678
6. feature 94: 0.008822020226963349
7. feature 79: 0.00872048865547228
8. feature 89: 0.008393957575172837
9. feature 49: 0.00833328823498188
10. feature 12: 0.008242356321677316
11. feature 82: 0.008114625429010737
12. feature 51: 0.008104053552752165
13. feature 55: 0.008094847876625212
14. feature 66: 0.008064408438414555
15. feature 47: 0.00785300220608914
16. feature 17: 0.0077133341443931394
17. feature 87: 0.007494065077920688
18. feature 41: 0.007424822763282093
19. feature 43: 0.007354073249384186
20. feature 24: 0.007233824574872333
上記をグラフとして可視化したものは以下の通り。
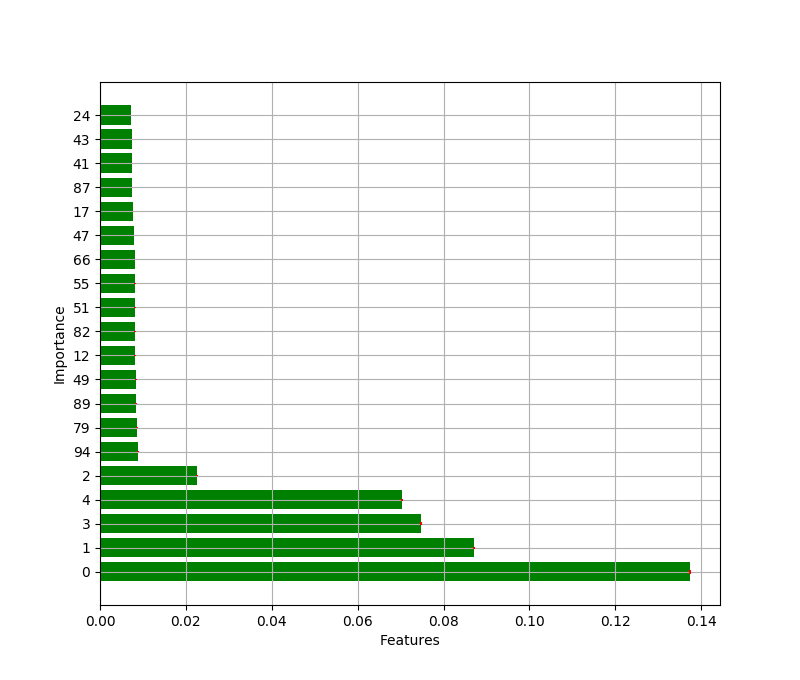
このように、モデルがその特徴量を重要と判断しているからといって、本当にそれが性能の向上に寄与するとは限らない。
RFE を使って特徴量を選択する
前置きが長くなったけど、そろそろ実際に RFE で特徴量を選択してみよう。
RFE は scikit-learn に実装がある。
scikit-learn.org
以下のサンプルコードでは RFE とランダムフォレストを使って 100 次元の特徴量から 5 次元を選択している。
また、選択した特徴量の重要度を出力すると共に、選択した特徴量についてホールドアウト検証で性能を確認している。
from sklearn.datasets import make_classification
from sklearn.ensemble import RandomForestClassifier
from sklearn.feature_selection import RFE
from sklearn.metrics import roc_auc_score
from sklearn.model_selection import train_test_split
from sklearn.model_selection import cross_val_predict
from sklearn.model_selection import StratifiedKFold
def main():
args = {
'n_samples': 1000,
'n_features': 100,
'n_informative': 5,
'n_redundant': 0,
'n_repeated': 0,
'n_classes': 2,
'random_state': 42,
'shuffle': False,
}
X, y = make_classification(**args)
clf = RandomForestClassifier(n_estimators=100,
random_state=42)
rfe = RFE(estimator=clf,
n_features_to_select=5,
verbose=1)
X_train, X_eval, y_train, y_eval = train_test_split(X, y,
shuffle=True,
random_state=42)
rfe.fit(X_eval, y_eval)
print('Feature ranking by RFF:', rfe.ranking_)
X_train_selected = X_train[:, rfe.support_]
skf = StratifiedKFold(n_splits=5,
shuffle=True,
random_state=42)
y_pred = cross_val_predict(clf, X_train_selected, y_train,
cv=skf,
method='predict_proba')
metric = roc_auc_score(y_train, y_pred[:, 1])
print('RFE selected features AUC:', metric)
if __name__ == '__main__':
main()
実行結果は次の通り。
意味がある特徴量である先頭の 5 次元がランキングで 1 位となっている。
どうやら、ちゃんと有効な特徴量が選択できているようだ。
$ python rfe.py
Fitting estimator with 100 features.
Fitting estimator with 99 features.
Fitting estimator with 98 features.
Fitting estimator with 97 features.
Fitting estimator with 96 features.
Fitting estimator with 95 features.
...(snip)...
Fitting estimator with 10 features.
Fitting estimator with 9 features.
Fitting estimator with 8 features.
Fitting estimator with 7 features.
Fitting estimator with 6 features.
Feature ranking by RFF: [ 1 1 1 1 1 59 14 44 79 61 58 67 70 41 77 33 51 24 3 34 9 71 63 91
73 47 53 26 86 17 13 94 88 31 2 62 19 25 11 48 95 84 56 10 68 18 74 45
39 15 43 96 32 80 52 36 4 57 82 50 27 78 69 85 93 65 29 7 30 55 46 49
20 60 72 16 42 92 23 64 81 22 38 90 66 28 35 76 83 8 37 21 54 40 87 75
5 12 6 89]
RFE selected features AUC: 0.9770972008875235
AUC が理想的な状況よりも落ちているのは、ホールドアウト検証なので使えるデータが減っているためだろうか。
選択する特徴量の数を最適化する
ところで、RFE では選択する特徴量の数をあらかじめ指定する必要がある。
先ほどのサンプルコードでは、あらかじめ有効な特徴量の数が分かっていたので 5 を指定した。
しかしながら、そもそもいくつ取り出すのが良いのかはあらかじめ分かっていない状況の方が多いだろう。
選択する特徴量の数は、一体どうやって決めれば良いのだろうか。
悩んだものの、そもそもハイパーパラメータの一種と捉えて最適化してしまえば良いのではと考えた。
そこで、続いては RFE で選択する特徴量の数を Optuna を使って最適化してみよう。
まずは Optuna をインストールしておく。
$ pip install optuna
以下のサンプルコードでは、AUC が最大になるように取り出す特徴量の数を最適化している。
from functools import partial
import optuna
from sklearn.datasets import make_classification
from sklearn.ensemble import RandomForestClassifier
from sklearn.feature_selection import RFE
from sklearn.metrics import roc_auc_score
from sklearn.model_selection import train_test_split
from sklearn.model_selection import cross_val_predict
from sklearn.model_selection import StratifiedKFold
def objective(X, y, trial):
"""最適化する目的関数"""
clf = RandomForestClassifier(n_estimators=100,
random_state=42)
n_features_to_select = trial.suggest_int('n_features_to_select', 1, 100),
rfe = RFE(estimator=clf,
n_features_to_select=n_features_to_select)
X_train, X_eval, y_train, y_eval = train_test_split(X, y,
shuffle=True,
random_state=42)
rfe.fit(X_eval, y_eval)
X_train_selected = X_train[:, rfe.support_]
skf = StratifiedKFold(n_splits=5,
shuffle=True,
random_state=42)
y_pred = cross_val_predict(clf, X_train_selected, y_train,
cv=skf,
method='predict_proba')
metric = roc_auc_score(y_train, y_pred[:, 1])
return metric
def main():
args = {
'n_samples': 500,
'n_features': 100,
'n_informative': 5,
'n_redundant': 0,
'n_repeated': 0,
'n_classes': 2,
'random_state': 42,
'shuffle': False,
}
X, y = make_classification(**args)
f = partial(objective, X, y)
study = optuna.create_study(direction='maximize')
study.optimize(f, n_trials=20)
print('params:', study.best_params)
if __name__ == '__main__':
main()
実行結果は次の通り。
ちゃんと選択すべき特徴量の数が 5 として最適化できた。
$ python rfeopt.py
[I 2019-04-20 19:42:22,874] Finished trial#0 resulted in value: 0.9571904165718188. Current best value is 0.9571904165718188 with parameters: {'n_features_to_select': 23}.
[I 2019-04-20 19:42:29,757] Finished trial#1 resulted in value: 0.9299169132711131. Current best value is 0.9571904165718188 with parameters: {'n_features_to_select': 23}.
[I 2019-04-20 19:42:35,810] Finished trial#2 resulted in value: 0.9395487138629638. Current best value is 0.9571904165718188 with parameters: {'n_features_to_select': 23}.
[I 2019-04-20 19:42:45,647] Finished trial#3 resulted in value: 0.8863390621443205. Current best value is 0.9571904165718188 with parameters: {'n_features_to_select': 23}.
[I 2019-04-20 19:42:56,298] Finished trial#4 resulted in value: 0.9714318233553381. Current best value is 0.9714318233553381 with parameters: {'n_features_to_select': 6}.
[I 2019-04-20 19:43:02,694] Finished trial#5 resulted in value: 0.9416543364443433. Current best value is 0.9714318233553381 with parameters: {'n_features_to_select': 6}.
[I 2019-04-20 19:43:04,807] Finished trial#6 resulted in value: 0.9038100386979285. Current best value is 0.9714318233553381 with parameters: {'n_features_to_select': 6}.
[I 2019-04-20 19:43:08,414] Finished trial#7 resulted in value: 0.9231874573184613. Current best value is 0.9714318233553381 with parameters: {'n_features_to_select': 6}.
[I 2019-04-20 19:43:14,815] Finished trial#8 resulted in value: 0.9392783974504895. Current best value is 0.9714318233553381 with parameters: {'n_features_to_select': 6}.
[I 2019-04-20 19:43:19,781] Finished trial#9 resulted in value: 0.9385954928295015. Current best value is 0.9714318233553381 with parameters: {'n_features_to_select': 6}.
[I 2019-04-20 19:43:27,921] Finished trial#10 resulted in value: 0.957503414523105. Current best value is 0.9714318233553381 with parameters: {'n_features_to_select': 6}.
[I 2019-04-20 19:43:37,352] Finished trial#11 resulted in value: 0.7167226269064422. Current best value is 0.9714318233553381 with parameters: {'n_features_to_select': 6}.
[I 2019-04-20 19:43:46,542] Finished trial#12 resulted in value: 0.9767385613475985. Current best value is 0.9767385613475985 with parameters: {'n_features_to_select': 5}.
[I 2019-04-20 19:43:55,495] Finished trial#13 resulted in value: 0.9558815160482587. Current best value is 0.9767385613475985 with parameters: {'n_features_to_select': 5}.
[I 2019-04-20 19:44:03,303] Finished trial#14 resulted in value: 0.9479854313680856. Current best value is 0.9767385613475985 with parameters: {'n_features_to_select': 5}.
[I 2019-04-20 19:44:12,028] Finished trial#15 resulted in value: 0.9614727976325973. Current best value is 0.9767385613475985 with parameters: {'n_features_to_select': 5}.
[I 2019-04-20 19:44:15,797] Finished trial#16 resulted in value: 0.9248235829729115. Current best value is 0.9767385613475985 with parameters: {'n_features_to_select': 5}.
[I 2019-04-20 19:44:16,820] Finished trial#17 resulted in value: 0.9016617345777374. Current best value is 0.9767385613475985 with parameters: {'n_features_to_select': 5}.
[I 2019-04-20 19:44:26,228] Finished trial#18 resulted in value: 0.8863390621443205. Current best value is 0.9767385613475985 with parameters: {'n_features_to_select': 5}.
[I 2019-04-20 19:44:33,167] Finished trial#19 resulted in value: 0.9437030503073071. Current best value is 0.9767385613475985 with parameters: {'n_features_to_select': 5}.
params: {'n_features_to_select': 5}
めでたしめでたし。