XGBoost (eXtreme Gradient Boosting) は勾配ブースティング決定木 (Gradient Boosting Decision Tree) のアルゴリズムを実装したオープンソースのライブラリ。
最近は、同じ GBDT 系のライブラリである LightGBM にややお株を奪われつつあるものの、依然として機械学習コンペティションの一つである Kaggle でよく使われている。
今回は、そんな XGBoost の Python バインディングを使ってみることにする。
使った環境は次の通り。
$ sw_vers
ProductName: Mac OS X
ProductVersion: 10.14.2
BuildVersion: 18C54
$ python -V
Python 3.7.2
もくじ
下準備
最初に、下準備として XGBoost と必要なパッケージを一通りインストールする。
そのために、まずは gcc をインストールしておく。
これは XGBoost が並列処理に OpenMP を使っている関係で必要になる。
$ brew install gcc@7
続いて XGBoost と、それ以外で今回使うパッケージをインストールする。
$ pip install xgboost scikit-learn matplotlib
乳がんデータセットを分類してみる
まずはハローワールド的な例として乳がんデータセットを使った二値分類 (Binary classification) から始める。
以下が XGBoost を使って乳がんデータセットを二値分類するサンプルコード。
なお、モデルの検証についてはここでの本題ではないことから、交差検証ではなくホールドアウト検証にとどめている。
最終的に検証用データで精度 (Accuracy) を確認している。
コードの説明についてはコメントを参照のこと。
import xgboost as xgb
import numpy as np
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
"""XGBoost で二値分類するサンプルコード"""
def main():
dataset = datasets.load_breast_cancer()
X, y = dataset.data, dataset.target
X_train, X_test, y_train, y_test = train_test_split(X, y,
test_size=0.3,
shuffle=True,
random_state=42,
stratify=y)
dtrain = xgb.DMatrix(X_train, label=y_train)
dtest = xgb.DMatrix(X_test, label=y_test)
xgb_params = {
'objective': 'binary:logistic',
'eval_metric': 'logloss',
}
bst = xgb.train(xgb_params,
dtrain,
num_boost_round=100,
)
y_pred_proba = bst.predict(dtest)
y_pred = np.where(y_pred_proba > 0.5, 1, 0)
acc = accuracy_score(y_test, y_pred)
print('Accuracy:', acc)
if __name__ == '__main__':
main()
上記のサンプルコードに適当な名前をつけて保存した上で実行する。
$ python xgbhelloworld.py
[22:52:44] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 18 extra nodes, 0 pruned nodes, max_depth=5
[22:52:44] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 14 extra nodes, 0 pruned nodes, max_depth=4
[22:52:44] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 14 extra nodes, 0 pruned nodes, max_depth=4
...(snip)...
[22:52:44] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 2 extra nodes, 0 pruned nodes, max_depth=1
[22:52:44] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 2 extra nodes, 0 pruned nodes, max_depth=1
[22:52:44] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 2 extra nodes, 0 pruned nodes, max_depth=1
Accuracy: 0.9649122807017544
ホールドアウト検証の結果、精度として 0.965 前後の値が得られた。
学習過程を可視化する
先ほどの例では、いつの間にか学習が終わってモデルができたという感じだった。
そこで、続いては学習が進む過程をグラフで可視化してみる。
次のサンプルコードでは、学習用データと検証用データに対する損失を折れ線グラフで出力する。
そのためには、まず evals オプションで学習用データと検証用データを渡す。
その上で evals_result オプションに過程を記録するための辞書を渡す。
学習ラウンド数 (num_boost_round) は先ほどよりも多く 1000 まで増やした。
import xgboost as xgb
import numpy as np
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
from matplotlib import pyplot as plt
"""XGBoost で学習の履歴を可視化するサンプルコード"""
def main():
dataset = datasets.load_breast_cancer()
X, y = dataset.data, dataset.target
X_train, X_test, y_train, y_test = train_test_split(X, y,
test_size=0.3,
shuffle=True,
random_state=42,
stratify=y)
dtrain = xgb.DMatrix(X_train, label=y_train)
dtest = xgb.DMatrix(X_test, label=y_test)
xgb_params = {
'objective': 'binary:logistic',
'eval_metric': 'logloss',
}
evals = [(dtrain, 'train'), (dtest, 'eval')]
evals_result = {}
bst = xgb.train(xgb_params,
dtrain,
num_boost_round=1000,
evals=evals,
evals_result=evals_result,
)
y_pred_proba = bst.predict(dtest)
y_pred = np.where(y_pred_proba > 0.5, 1, 0)
acc = accuracy_score(y_test, y_pred)
print('Accuracy:', acc)
train_metric = evals_result['train']['logloss']
plt.plot(train_metric, label='train logloss')
eval_metric = evals_result['eval']['logloss']
plt.plot(eval_metric, label='eval logloss')
plt.grid()
plt.legend()
plt.xlabel('rounds')
plt.ylabel('logloss')
plt.show()
if __name__ == '__main__':
main()
先ほどと同じように、適当な名前をつけて実行する。
$ python xgbhistory.py
...(snip)...
Accuracy: 0.9649122807017544
すると、次のようなグラフが得られる。
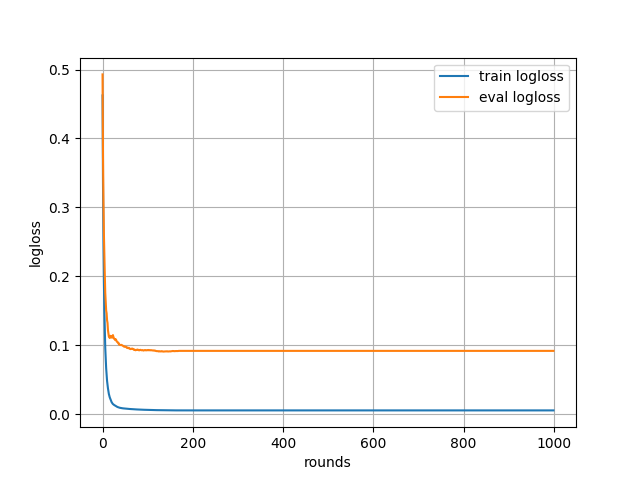
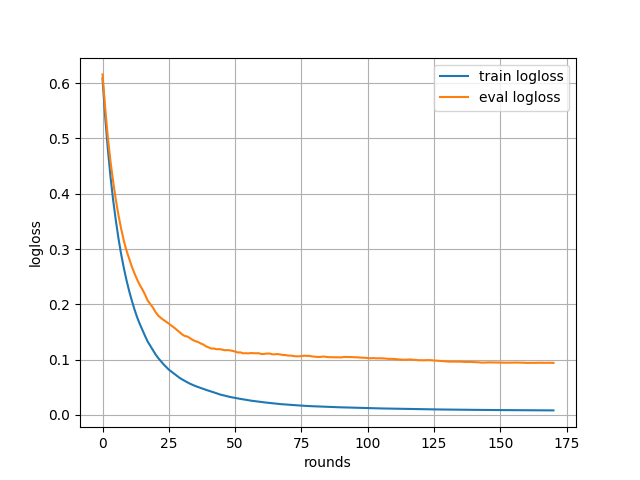
学習が進むにつれて学習用データと検証用データの損失が減っていくことが分かる。
とはいえ、過学習はしていないようだけど 100 ラウンド前後で損失の減少は止まっているように見える。
損失が減らなくなったら学習を打ち切る
先ほどの例で分かる通り、損失が減らなくなったらそれ以上学習する必要はない。
それ以上回してしまうと、下手をすると損失が増える (過学習) することも考えられる。
そこで、続いては損失が減らなくなったタイミングで学習を打ち切るようにしてみる。
次のサンプルコードでは early_stopping_rounds オプションを指定することでそれを実現している。
このオプションを使うと、指定したラウンド数の間で損失が減らなかったときに学習を打ち切れる。
import xgboost as xgb
import numpy as np
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
from matplotlib import pyplot as plt
"""XGBoost で early_stopping_rounds を使って学習ラウンド数を最適化するサンプルコード"""
def main():
dataset = datasets.load_breast_cancer()
X, y = dataset.data, dataset.target
X_train, X_test, y_train, y_test = train_test_split(X, y,
test_size=0.3,
shuffle=True,
random_state=42,
stratify=y)
dtrain = xgb.DMatrix(X_train, label=y_train)
dtest = xgb.DMatrix(X_test, label=y_test)
xgb_params = {
'objective': 'binary:logistic',
'eval_metric': 'logloss',
}
evals = [(dtrain, 'train'), (dtest, 'eval')]
evals_result = {}
bst = xgb.train(xgb_params,
dtrain,
num_boost_round=1000,
early_stopping_rounds=10,
evals=evals,
evals_result=evals_result,
)
y_pred_proba = bst.predict(dtest)
y_pred = np.where(y_pred_proba > 0.5, 1, 0)
acc = accuracy_score(y_test, y_pred)
print('Accuracy:', acc)
train_metric = evals_result['train']['logloss']
plt.plot(train_metric, label='train logloss')
eval_metric = evals_result['eval']['logloss']
plt.plot(eval_metric, label='eval logloss')
plt.grid()
plt.legend()
plt.xlabel('rounds')
plt.ylabel('logloss')
plt.show()
if __name__ == '__main__':
main()
上記を実行してみよう。
ログ出力から、検証用データの損失に対して early stopping がかかるようになっていることが分かる。
$ python xgbearlystop.py
[23:05:43] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 18 extra nodes, 0 pruned nodes, max_depth=5
[0] train-logloss:0.462396 eval-logloss:0.492899
Multiple eval metrics have been passed: 'eval-logloss' will be used for early stopping.
Will train until eval-logloss hasn't improved in 10 rounds.
[23:05:43] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 14 extra nodes, 0 pruned nodes, max_depth=4
...(snip)...
[23:05:44] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 2 extra nodes, 0 pruned nodes, max_depth=1
[100] train-logloss:0.006161 eval-logloss:0.09275
[23:05:44] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 2 extra nodes, 0 pruned nodes, max_depth=1
[101] train-logloss:0.00614 eval-logloss:0.093094
Stopping. Best iteration:
[91] train-logloss:0.00637 eval-logloss:0.092265
Accuracy: 0.9649122807017544
今度は 101 ラウンドまでしか学習が進んでいない。
また、その中でも最も損失の少なかった 91 ラウンド目が結果として使われたことが分かる。
学習過程のグラフは次のようなものが得られた。
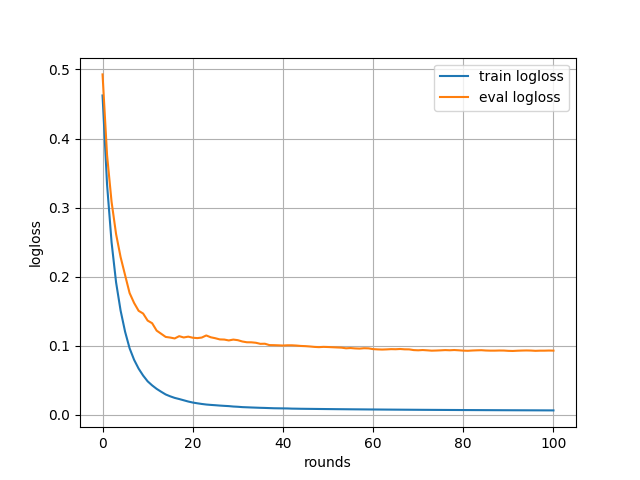
たしかに 100 前後の学習ラウンドまでしか表示されていない。
scikit-learn インターフェースを使ってみる
XGBoost には、ネイティブな API の他に scikit-learn 互換の API を持ったインターフェースもある。
続いては、これを使ってみよう。
次のサンプルコードでは scikit-learn 互換の API を備えた XGBClassifier を使っている。
ただし、実現していることは先ほどと全く変わらない。
import xgboost as xgb
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
from matplotlib import pyplot as plt
"""XGBoost の scikit-learn インターフェースを使ったサンプルコード (二値分類)"""
def main():
dataset = datasets.load_breast_cancer()
X, y = dataset.data, dataset.target
X_train, X_test, y_train, y_test = train_test_split(X, y,
test_size=0.3,
shuffle=True,
random_state=42,
stratify=y)
clf = xgb.XGBClassifier(objective='binary:logistic',
n_estimators=1000)
evals_result = {}
clf.fit(X_train, y_train,
eval_metric='logloss',
eval_set=[
(X_train, y_train),
(X_test, y_test),
],
early_stopping_rounds=10,
callbacks=[
xgb.callback.record_evaluation(evals_result)
],
)
y_pred = clf.predict(X_test)
acc = accuracy_score(y_test, y_pred)
print('Accuracy:', acc)
train_metric = evals_result['validation_0']['logloss']
plt.plot(train_metric, label='train logloss')
eval_metric = evals_result['validation_1']['logloss']
plt.plot(eval_metric, label='eval logloss')
plt.grid()
plt.legend()
plt.xlabel('rounds')
plt.ylabel('logloss')
plt.show()
if __name__ == '__main__':
main()
上記を実行してみよう。
$ python xgbscikit.py
[0] validation_0-logloss:0.607962 validation_1-logloss:0.615844
Multiple eval metrics have been passed: 'validation_1-logloss' will be used for early stopping.
Will train until validation_1-logloss hasn't improved in 10 rounds.
[1] validation_0-logloss:0.538811 validation_1-logloss:0.555234
[2] validation_0-logloss:0.480826 validation_1-logloss:0.5015
[3] validation_0-logloss:0.430842 validation_1-logloss:0.458286
...(snip)...
[170] validation_0-logloss:0.008248 validation_1-logloss:0.094108
Stopping. Best iteration:
[160] validation_0-logloss:0.008465 validation_1-logloss:0.094088
Accuracy: 0.9649122807017544
学習ラウンド数は先ほどと違っているものの、最終的に得られた精度は変わっていないようだ。
学習過程のグラフは次のようなものが得られた。
多値分類問題を扱う
続いてはタスク設定として多値分類 (Multiclass classification) を試してみよう。
この場合、二値分類とは学習するときのパラメータが異なる。
次のサンプルコードでは XGBoost で Iris データセットを使った多値分類問題を扱っている。
import xgboost as xgb
from matplotlib import pyplot as plt
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
"""XGBoost で多値分類するサンプルコード"""
def main():
dataset = datasets.load_iris()
X, y = dataset.data, dataset.target
X_train, X_test, y_train, y_test = train_test_split(X, y,
test_size=0.3,
shuffle=True,
random_state=42,
stratify=y)
dtrain = xgb.DMatrix(X_train, label=y_train)
dtest = xgb.DMatrix(X_test, label=y_test)
xgb_params = {
'objective': 'multi:softmax',
'num_class': 3,
'eval_metric': 'mlogloss',
}
evals = [(dtrain, 'train'), (dtest, 'eval')]
evals_result = {}
bst = xgb.train(xgb_params,
dtrain,
num_boost_round=1000,
early_stopping_rounds=10,
evals=evals,
evals_result=evals_result,
)
y_pred = bst.predict(dtest)
acc = accuracy_score(y_test, y_pred)
print('Accuracy:', acc)
train_metric = evals_result['train']['mlogloss']
plt.plot(train_metric, label='train logloss')
eval_metric = evals_result['eval']['mlogloss']
plt.plot(eval_metric, label='eval logloss')
plt.grid()
plt.legend()
plt.xlabel('rounds')
plt.ylabel('logloss')
plt.show()
if __name__ == '__main__':
main()
上記を実行してみよう。
$ python xgbmulticlass.py
[23:14:48] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 2 extra nodes, 0 pruned nodes, max_depth=1
[23:14:48] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 8 extra nodes, 0 pruned nodes, max_depth=3
[23:14:48] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 4 extra nodes, 0 pruned nodes, max_depth=2
[0] train-mlogloss:0.742287 eval-mlogloss:0.765776
Multiple eval metrics have been passed: 'eval-mlogloss' will be used for early stopping.
Will train until eval-mlogloss hasn't improved in 10 rounds.
...
Stopping. Best iteration:
[16] train-mlogloss:0.039852 eval-mlogloss:0.195911
Accuracy: 0.9333333333333333
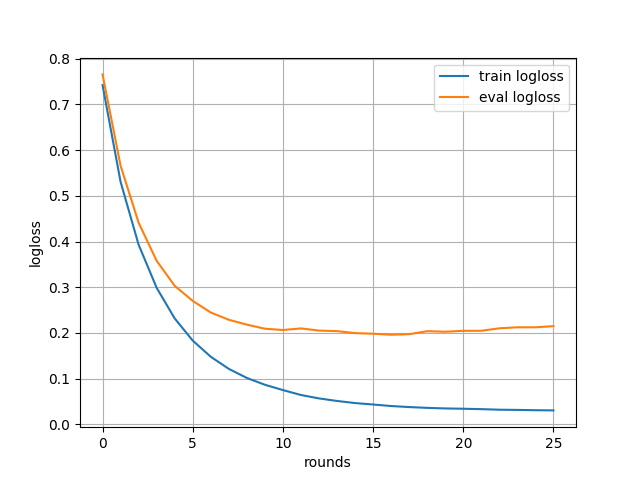
乳がんデータセットに比べると、単純な分だけラウンド数がかなり少ないようだ。
得られた学習過程のグラフは次の通り。
学習用データの損失は減っているものの、検証用データの損失が減らない状況が生じていることから過学習の予兆が見られる。
特徴量の重要度を可視化する
XGBoost は決定木の仲間ということで特徴量の重要度 (Feature Importance) を可視化する機能を備えている。
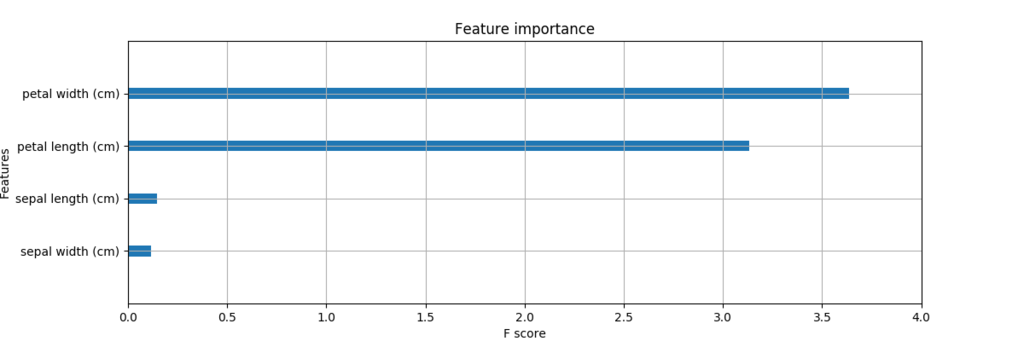
次のサンプルコードでは、Iris データセットの分類にどの特徴量が有効だったのかを性能のゲインにもとづいて可視化している。
import xgboost as xgb
from sklearn import datasets
from sklearn.model_selection import train_test_split
from matplotlib import pyplot as plt
"""XGBoost で特徴量の重要度を可視化するサンプルコード"""
def main():
dataset = datasets.load_iris()
X, y = dataset.data, dataset.target
X_train, X_test, y_train, y_test = train_test_split(X, y,
test_size=0.3,
shuffle=True,
random_state=42,
stratify=y)
dtrain = xgb.DMatrix(X_train, label=y_train,
feature_names=dataset.feature_names)
dtest = xgb.DMatrix(X_test, label=y_test,
feature_names=dataset.feature_names)
xgb_params = {
'objective': 'multi:softmax',
'num_class': 3,
'eval_metric': 'mlogloss',
}
evals = [(dtrain, 'train'), (dtest, 'eval')]
evals_result = {}
bst = xgb.train(xgb_params,
dtrain,
num_boost_round=1000,
early_stopping_rounds=10,
evals=evals,
evals_result=evals_result,
)
_, ax = plt.subplots(figsize=(12, 4))
xgb.plot_importance(bst,
ax=ax,
importance_type='gain',
show_values=False)
plt.show()
if __name__ == '__main__':
main()
上記を実行してみよう。
$ python xgbfeatimp.py
[23:19:37] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 2 extra nodes, 0 pruned nodes, max_depth=1
[23:19:37] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 8 extra nodes, 0 pruned nodes, max_depth=3
[23:19:37] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 4 extra nodes, 0 pruned nodes, max_depth=2
[0] train-mlogloss:0.742287 eval-mlogloss:0.765776
Multiple eval metrics have been passed: 'eval-mlogloss' will be used for early stopping.
Will train until eval-mlogloss hasn't improved in 10 rounds.
...(snip)...
Stopping. Best iteration:
[16] train-mlogloss:0.039852 eval-mlogloss:0.195911
次のようなグラフが得られる。
どうやら petal length と petal width が分類する上で有効なようだ。
回帰問題を扱ってみる
続いては XGBoost で回帰問題を扱ってみる。
回帰問題を扱うときは学習時のパラメータとして渡す objective が reg から始まるようになる。
次のサンプルコードでは XGBoost で Boston データセットを回帰している。
学習と検証の評価指標には RMSE (Root Mean Squared Error) を用いた。
import math
import xgboost as xgb
from matplotlib import pyplot as plt
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn.metrics import mean_squared_error
"""XGBoost で回帰するサンプルコード"""
def main():
dataset = datasets.load_boston()
X, y = dataset.data, dataset.target
X_train, X_test, y_train, y_test = train_test_split(X, y,
test_size=0.3,
shuffle=True,
random_state=42,
)
dtrain = xgb.DMatrix(X_train, label=y_train)
dtest = xgb.DMatrix(X_test, label=y_test)
xgb_params = {
'objective': 'reg:linear',
'eval_metric': 'rmse',
}
evals = [(dtrain, 'train'), (dtest, 'eval')]
evals_result = {}
bst = xgb.train(xgb_params,
dtrain,
num_boost_round=1000,
early_stopping_rounds=10,
evals=evals,
evals_result=evals_result,
)
y_pred = bst.predict(dtest)
mse = mean_squared_error(y_test, y_pred)
print('RMSE:', math.sqrt(mse))
train_metric = evals_result['train']['rmse']
plt.plot(train_metric, label='train rmse')
eval_metric = evals_result['eval']['rmse']
plt.plot(eval_metric, label='eval rmse')
plt.grid()
plt.legend()
plt.xlabel('rounds')
plt.ylabel('rmse')
plt.show()
if __name__ == '__main__':
main()
上記を実行してみよう。
$ python xgbreg.py
[23:25:09] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 22 extra nodes, 0 pruned nodes, max_depth=5
[0] train-rmse:17.5096 eval-rmse:16.1546
Multiple eval metrics have been passed: 'eval-rmse' will be used for early stopping.
Will train until eval-rmse hasn't improved in 10 rounds.
...(snip)...
Stopping. Best iteration:
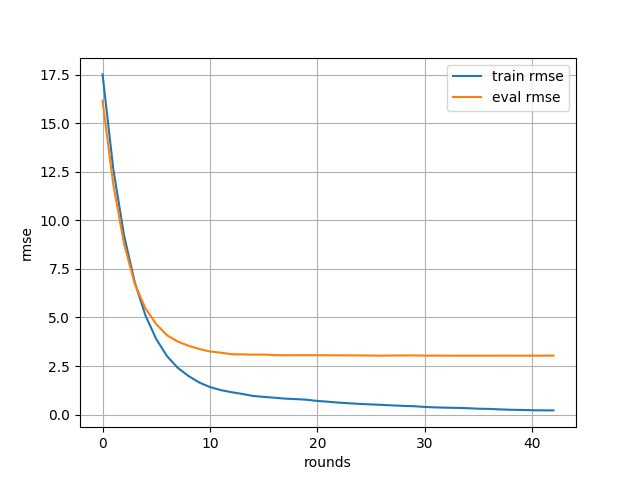
[33] train-rmse:0.344815 eval-rmse:3.03055
RMSE: 3.0332711348600068
学習過程の損失をプロットしたグラフは次のようになった。
カスタムメトリックを扱う
これまでは XGBoost に組み込みで入っていた評価指標を用いて学習の進み具合を評価していた。
続いては自分で書いたカスタムメトリックを使って学習してみる。
次のサンプルコードでは、再び乳がんデータセットを使った二値分類を扱っている。
ただし、学習を評価するメトリックとして精度 (Accuracy) を計測するカスタムメトリックを使っている。
カスタムメトリックを扱うにはオプションとして feval に自分で書いた関数を渡す。
カスタムメトリックの関数は、評価指標の名前と値をタプルまたはリストで返すように作る。
import xgboost as xgb
import numpy as np
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
from matplotlib import pyplot as plt
"""XGBoost で学習時にカスタムメトリックを使ったサンプルコード"""
def feval_accuracy(pred_proba, dtrain):
"""カスタムメトリックを計算する関数"""
y_true = dtrain.get_label().astype(int)
y_pred = np.where(pred_proba > 0.5, 1, 0)
acc = accuracy_score(y_true, y_pred)
return 'accuracy', 1 - acc
def main():
dataset = datasets.load_breast_cancer()
X, y = dataset.data, dataset.target
X_train, X_test, y_train, y_test = train_test_split(X, y,
test_size=0.3,
shuffle=True,
random_state=42,
stratify=y)
dtrain = xgb.DMatrix(X_train, label=y_train)
dtest = xgb.DMatrix(X_test, label=y_test)
xgb_params = {
'objective': 'binary:logistic',
'eval_metric': 'logloss',
}
evals = [(dtrain, 'train'), (dtest, 'eval')]
evals_result = {}
bst = xgb.train(xgb_params,
dtrain,
num_boost_round=1000,
early_stopping_rounds=10,
evals=evals,
evals_result=evals_result,
feval=feval_accuracy,
)
y_pred_proba = bst.predict(dtest)
y_pred = np.where(y_pred_proba > 0.5, 1, 0)
acc = accuracy_score(y_test, y_pred)
print('Accuracy:', acc)
_, ax1 = plt.subplots(figsize=(8, 4))
train_metric = evals_result['train']['logloss']
ax1.plot(train_metric, label='train logloss', c='r')
eval_metric = evals_result['eval']['logloss']
ax1.plot(eval_metric, label='eval logloss', c='g')
ax1.set_ylabel('logloss')
ax1.legend()
ax1.set_xlabel('rounds')
ax2 = ax1.twinx()
eval_custom_metric = evals_result['eval']['accuracy']
ax2.plot(eval_custom_metric, label='eval accuracy', c='b')
ax2.set_ylabel('accuracy')
ax2.legend()
plt.grid()
plt.show()
if __name__ == '__main__':
main()
上記を実行してみる。
$ python xgbcustmetric.py
[23:35:00] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 18 extra nodes, 0 pruned nodes, max_depth=5
[0] train-logloss:0.462396 eval-logloss:0.492899 train-accuracy:0.017588 eval-accuracy:0.070175
Multiple eval metrics have been passed: 'eval-accuracy' will be used for early stopping.
Will train until eval-accuracy hasn't improved in 10 rounds.
...(snip)...
Stopping. Best iteration:
[14] train-logloss:0.029256 eval-logloss:0.112603 train-accuracy:0.005025 eval-accuracy:0.046784
Accuracy: 0.9532163742690059
得られた学習過程のグラフは次の通り。
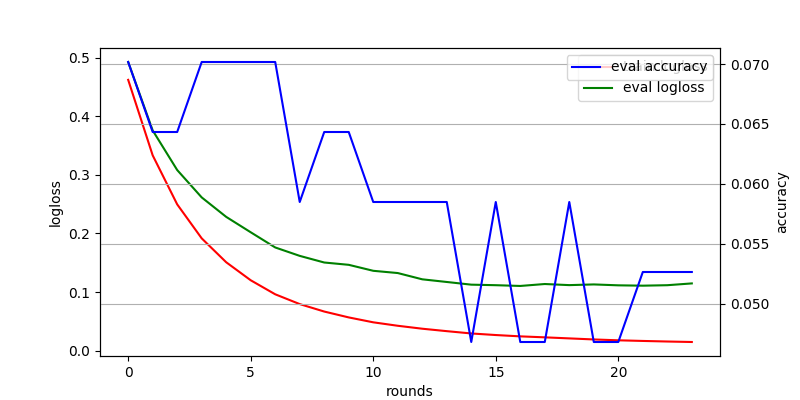
学習の評価指標として精度 (Accuracy) を使うと、早々に学習が打ち切られてしまっているようだ。
そのため、最終的に得られた精度も低いものになってしまっている。
これは、おそらく一つの要素が正しく分類できたか・できなかったに結果が大きく左右されてしまうため。
まあ、結果はそんなに良くないけど、こんな感じで書けるよという例として。
組み込みの交差検証の機能を使ってみる
XGBoost には、組み込みの交差検証 (Cross Validation) の機能がある。
正直、そんなに使いやすいものではないけど、とりあえずあるよということで紹介しておく。
次のサンプルコードでは、XGBoost に組み込みで入っている交差検証の機能を使っている。
機能は xgboost.cv() という関数が起点になる。
基本的なパラメータについては xgboost.train() で普通に学習するときとさほど変わらない。
関数から最終的に得られるのは、学習過程におけるメトリックの値の変化。
import xgboost as xgb
import numpy as np
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
from matplotlib import pyplot as plt
"""XGBoost 組み込みの交差検証を使ったサンプルコード"""
def main():
dataset = datasets.load_breast_cancer()
X, y = dataset.data, dataset.target
dtrain = xgb.DMatrix(X, label=y)
xgb_params = {
'objective': 'binary:logistic',
'eval_metric': 'logloss',
}
history = xgb.cv(xgb_params,
dtrain,
num_boost_round=1000,
early_stopping_rounds=10,
nfold=10,
stratified=True,
verbose_eval=True,
)
train_metric = history['train-logloss-mean']
plt.plot(train_metric, label='train logloss')
eval_metric = history['test-logloss-mean']
plt.plot(eval_metric, label='eval logloss')
plt.grid()
plt.legend()
plt.xlabel('rounds')
plt.ylabel('logloss')
plt.show()
if __name__ == '__main__':
main()
上記を実行してみる。
$ python xgbcustmetric.py
[23:35:00] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 18 extra nodes, 0 pruned nodes, max_depth=5
[0] train-logloss:0.462396 eval-logloss:0.492899 train-accuracy:0.017588 eval-accuracy:0.070175
Multiple eval metrics have been passed: 'eval-accuracy' will be used for early stopping.
Will train until eval-accuracy hasn't improved in 10 rounds.
...(snip)...
[23:39:47] src/tree/updater_prune.cc:74: tree pruning end, 1 roots, 2 extra nodes, 0 pruned nodes, max_depth=1
すると、次のような学習過程のグラフが得られる。
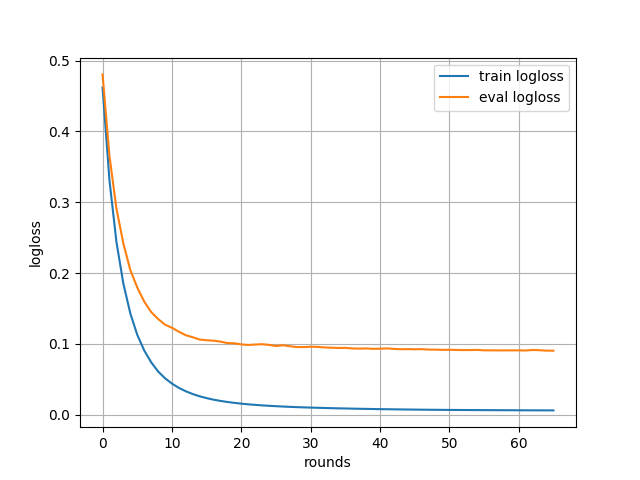
いじょう。
まとめ
今回は XGBoost の機能を色々と試してみた。