機械学習モデルにおいて、人間によるチューニングが必要なパラメータをハイパーパラメータと呼ぶ。
ハイパーパラメータをチューニングするやり方は色々とある。
例えば、良さそうなパラメータの組み合わせを全て試すグリッドサーチや、無作為に試すランダムサーチなど。
今回は、それとはちょっと違ったベイズ最適化というやり方を試してみる。
ベイズ最適化では、過去の試行結果から次に何処を調べれば良いかを確率分布と獲得関数にもとづいて決める。
これにより、比較的少ない試行回数でより優れたハイパーパラメータが選べるとされる。
Python でベイズ最適化をするためのパッケージとしては Bayesian Optimization や skopt、GPyOpt などがある。
今回は、その中でも Bayesian Optimization を使ってみることにした。
使った環境は次の通り。
$ sw_vers
ProductName: Mac OS X
ProductVersion: 10.13.6
BuildVersion: 17G65
$ python -V
Python 3.6.6
下準備
まずは今回使う Python のパッケージを一通りインストールしておく。
$ pip install bayesian-optimization matplotlib tqdm
基本的な考え方
最初に、単純な例を使って基本的な考え方を説明しておきたい。
まず、機械学習モデルにおけるハイパーパラメータの選択は、未知の関数の出力を最大化あるいは最小化する問題と捉えられる。
これは、ハイパーパラメータを入力として、それで学習した機械学習モデルを関数、モデルを評価して得られた何らかの性能指標 (出力) を最も良くする (最大化・最小化) ことが目的のため。
つまり、未知の関数の出力が最大あるいは最小となる点を見つけることができるなら、それはハイパーパラメータの選択にも適用できる可能性がある。
ごく単純な例として、次のような関数の出力を最大化することを考えてみよう。
グラフからは、だいたい x が 2 あたりで y が最大の 1.4 前後となることが分かる。
それ以外の場所では、だいたい 0 と 6 あたりに局所解が存在していて、そこでは y が 1 くらいになる。

ただし、この関数はあくまで本来は未知で、上記のグラフはあらかじめ分からないという前提になる。
その状況で、どうやら x が 2 あたりで y が最大になるっぽいぞ、ということを知りたい。
ちなみに、このグラフは次のようなコードで作った。
この中にある関数 f() が未知の関数ということになる。
import math
import numpy as np
from matplotlib import pyplot as plt
def f(x):
"""最大値を見つけたい関数"""
return math.exp(-(x - 2) ** 2) + math.exp(-(x - 6) ** 2 / 10) + 1 / (x ** 2 + 1)
def main():
X = [x for x in np.arange(-5, 15, 0.1)]
y = [f(x) for x in X]
plt.plot(X, y)
plt.xlabel('x')
plt.ylabel('y')
plt.grid()
plt.show()
if __name__ == '__main__':
main()
上記を適当な名前で保存して実行すると、先ほどのグラフが得られる。
$ python fplot.py
ベイズ最適化で関数の最大値を見つける
先ほどの関数を例にして、実際に Bayesian Optimization を使って関数が最大となる場所を探してみよう。
早速、以下にサンプルコードを示す。
大体の使い方はコメントを見れば分かると思う。
基本的には、探すパラメータとその範囲、そして初期点と探索するイテレーションの数を指定するだけで良い。
import math
from bayes_opt import BayesianOptimization
def f(x):
"""最大値を見つけたい関数"""
return math.exp(-(x - 2) ** 2) + math.exp(-(x - 6) ** 2 / 10) + 1 / (x ** 2 + 1)
def main():
pbounds = {
'x': (-5, 15),
}
bo = BayesianOptimization(f=f, pbounds=pbounds)
bo.maximize(init_points=3, n_iter=10)
print(bo.res['max'])
if __name__ == '__main__':
main()
実行結果は次の通り。
尚、結果は実行する毎に違ったものになる。
試行によっては局所解を答えてしまう場合もあるかも。
$ python bo.py
Initialization
-----------------------------------------
Step | Time | Value | x |
1 | 00m00s | 0.87632 | 4.6393 |
2 | 00m00s | 0.12396 | -2.6651 |
3 | 00m00s | 0.75934 | -0.5850 |
Bayesian Optimization
-----------------------------------------
Step | Time | Value | x |
4 | 00m00s | 0.00520 | 14.6498 |
5 | 00m00s | 0.39154 | 9.1121 |
6 | 00m00s | 1.39524 | 2.0885 |
7 | 00m00s | 1.17158 | 1.4247 |
8 | 00m00s | 0.80735 | 3.1369 |
9 | 00m00s | 0.97558 | 6.6884 |
10 | 00m00s | 0.04176 | 11.7992 |
11 | 00m00s | 0.03847 | -5.0000 |
12 | 00m00s | 1.02148 | 5.7120 |
13 | 00m00s | 0.74644 | 7.7734 |
{'max_val': 1.3952415083439782, 'max_params': {'x': 2.0884969890991476}}
上記の試行では、関数が最大となる場所は x が 2.08 あたりで、値は 1.39 くらいという結果が得られた。
先ほどグラフから目視で読み取った内容と整合している。
試行の過程を可視化してみる
先ほどの例では何となく上手くいくことは分かった。
ただ、ベイズ最適化が具体的に何処をどう探索しているのかよく分からない。
そこで、続いてはその過程を可視化してみることにする。
次のサンプルコードでは、ベイズ最適化の過程をグラフとしてプロットしている。
import math
import numpy as np
from bayes_opt import BayesianOptimization
from matplotlib import pyplot as plt
def f(x):
"""最大値を見つけたい関数"""
return math.exp(-(x - 2) ** 2) + math.exp(-(x - 6) ** 2 / 10) + 1 / (x ** 2 + 1)
def plot_bo(bo):
X = [x for x in np.arange(-5, 15, 0.1)]
y = [f(x) for x in X]
plt.plot(X, y, label='true')
xs = [p['x'] for p in bo.res['all']['params']]
ys = bo.res['all']['values']
plt.scatter(xs, ys, c='green', s=20, zorder=10, label='sample')
mean, sigma = bo.gp.predict(np.array(X).reshape(-1, 1), return_std=True)
plt.plot(X, mean, label='pred')
plt.fill_between(X, mean + sigma, mean - sigma, alpha=0.1)
max_x = bo.res['max']['max_params']['x']
max_y = bo.res['max']['max_val']
plt.scatter([max_x], [max_y], c='red', s=50, zorder=10, label='pred_max')
def main():
pbounds = {
'x': (-5, 15),
}
bo = BayesianOptimization(f=f, pbounds=pbounds)
bo.maximize(init_points=3, n_iter=10)
plot_bo(bo)
plt.legend()
plt.grid()
plt.show()
if __name__ == '__main__':
main()
適当な名前をつけて実行してみよう。
$ python boplot.py
Initialization
-----------------------------------------
Step | Time | Value | x |
1 | 00m00s | 0.06077 | -3.9330 |
2 | 00m00s | 0.74088 | 7.7945 |
3 | 00m00s | 0.12176 | 10.6689 |
Bayesian Optimization
-----------------------------------------
Step | Time | Value | x |
4 | 00m00s | 1.12790 | 2.6188 |
5 | 00m00s | 0.87412 | 4.6285 |
6 | 00m00s | 1.04842 | 0.0374 |
7 | 00m01s | 1.14774 | 1.3868 |
8 | 00m01s | 1.39986 | 1.9524 |
9 | 00m00s | 0.00473 | 15.0000 |
10 | 00m00s | 0.26949 | -1.6584 |
11 | 00m00s | 1.02154 | 6.1962 |
12 | 00m00s | 0.01546 | 12.8301 |
13 | 00m00s | 0.39699 | 9.0894 |
すると、例えば以下のようなグラフが得られる。
ここで true となっているのが真の関数で pred がベイズ最適化で推定した関数となる。
推定するのに用いた点は sample で、見つけた最大値と思われる場所は pred_max で図示している。
網掛けになっているのは、その周辺を調べていないことから、まだ推定にバラつきが大きいことを示している。

上記を見ると、いくつかの点を調べることで的確に真の関数に近いものを推定し、最大となる箇所を見つけていることが分かる。
獲得関数 (Acquisition Function)
ベイズ最適化では、次に何処を探すのかを確率分布にもとづいて獲得関数が決める。
使う獲得関数によって、まだ調べていない場所の探索に重きを置くのか、それとも着実な改善が見込める場所を探すのか、といった味付けが変わる。
Bayesian Optimization で使える獲得関数は以下の三つで、デフォルトは "ucb" になっている。
- Upper Confidence Bound (ucb)
- Probability of Improvement (poi)
- Expected Improvement (ei)
ちなみに Probability of Improvement は局所解に陥りやすいため、残り二つのどちらかを使うのが良さそう。
以下のサンプルコードでは、それぞれの獲得関数がどのように試行するのかをグラフに可視化してみた。
獲得関数のパラメータも二種類ずつ設定している。
import math
import numpy as np
from bayes_opt import BayesianOptimization
from matplotlib import pyplot as plt
from tqdm import tqdm
def f(x):
"""最大値を見つけたい関数"""
return math.exp(-(x - 2) ** 2) + math.exp(-(x - 6) ** 2 / 10) + 1 / (x ** 2 + 1)
def plot_bo(bo):
X = [x for x in np.arange(-5, 15, 0.1)]
y = [f(x) for x in X]
plt.plot(X, y, label='true')
xs = [p['x'] for p in bo.res['all']['params']]
ys = bo.res['all']['values']
plt.scatter(xs, ys, c='green', s=20, zorder=10, label='sample')
mean, sigma = bo.gp.predict(np.array(X).reshape(-1, 1), return_std=True)
plt.plot(X, mean, label='pred')
plt.fill_between(X, mean + sigma, mean - sigma, alpha=0.1)
max_x = bo.res['max']['max_params']['x']
max_y = bo.res['max']['max_val']
plt.scatter([max_x], [max_y], c='red', s=50, zorder=10, label='pred_max')
def main():
pbounds = {
'x': (-5, 15),
}
acq_params = [
('ucb_kappa_1', {
'acq': 'ucb',
'kappa': 1,
}),
('ucb_kappa_10', {
'acq': 'ucb',
'kappa': 10,
}),
('ei_xi_1e-4', {
'acq': 'ei',
'xi': 1e-4,
}),
('ei_xi_1e-1', {
'acq': 'ei',
'xi': 1e-1,
}),
('poi_xi_1e-4', {
'acq': 'poi',
'xi': 1e-4,
}),
('poi_xi_1e-1', {
'acq': 'poi',
'xi': 1e-1,
}),
]
plt.figure(figsize=(8, 12))
for i, (name, acq_param) in tqdm(list(enumerate(acq_params))):
bo = BayesianOptimization(f=f, pbounds=pbounds, verbose=0)
bo.maximize(init_points=3, n_iter=10, **acq_param)
plt.subplot(3, 2, i + 1)
plt.title(name)
plot_bo(bo)
plt.show()
if __name__ == '__main__':
main()
パラメータは、まだちゃんと理解できていないものの、次のように解釈できるっぽい。
- kappa
- 獲得関数が "ucb" のときに有効なパラメータ
- 大きくするほど「探索」に重きを置く
- xi
- 獲得関数が "poi" と "ei" で有効なパラメータ
- 大きくするほど既知の最大値から改善が見込める幅が大きな場所を探す
上記を適当な名前で保存して実行しよう。
終わるのには結構時間がかかるし、いくらか警告が出るかもしれない。
$ python boacq.py
...
すると、次のようなグラフが得られる。

獲得関数が "poi" でパラメータの "xi" が 1e-4 のパターンでは局所解に陥っていることが分かる。
機械学習モデルにベイズ最適化を適用する
だいぶ回り道したけど、ここからが今回の本題になる。
続いては、ベイズ最適化を使ってサポートベクターマシンのハイパーパラメータを選んでみよう。
RBF カーネルのサポートベクターマシンには "C" と "gamma" という二つのハイパーパラメータがある。
これをベイズ最適化を使って選ぶことにする。
以下にサンプルコードを示す。
基本的にやっていることは先ほどと変わらない。
まず、返り値を最大化したい関数を f() として用意している。
この関数は、渡されたハイパーパラメータを使ってサポートベクターマシンを学習する。
そして、学習したモデルを 3-Fold CV を使って精度 (Accuracy) で評価して、各結果の平均を返すことになる。
あとは、その関数の精度がなるべく高くなるようにベイズ最適化でハイパーパラメータを選ぶ。
import functools
from sklearn.svm import SVC
from sklearn.model_selection import KFold
from sklearn.model_selection import cross_val_score
from sklearn import datasets
import numpy as np
from bayes_opt import BayesianOptimization
from matplotlib import pyplot as plt
from matplotlib import cm
def f(X, y, **params):
"""最大化したい関数 (モデルを交差検証して得たスコアを返す)"""
svm = SVC(kernel='rbf', **params)
kf = KFold(n_splits=4, shuffle=True, random_state=42)
scores = cross_val_score(svm, X=X, y=y, cv=kf)
return scores.mean()
def main():
dataset = datasets.load_iris()
X, y = dataset.data, dataset.target
pf = functools.partial(f, X, y)
pbounds = {
'C': (1e+0, 1e+2),
'gamma': (1e-2, 1e+1),
}
bo = BayesianOptimization(f=pf, pbounds=pbounds)
bo.maximize(init_points=3, n_iter=20)
print(bo.res['max'])
xs = [p['C'] for p in bo.res['all']['params']]
ys = [p['gamma'] for p in bo.res['all']['params']]
zs = np.array(bo.res['all']['values'])
s_zs = (zs - zs.min()) / (zs.max() - zs.min())
sc = plt.scatter(xs, ys, c=s_zs, s=20, zorder=10, cmap=cm.cool)
plt.colorbar(sc)
plt.xlabel('C')
plt.xscale('log')
plt.ylabel('gamma')
plt.yscale('log')
plt.grid()
plt.show()
if __name__ == '__main__':
main()
上記に適当な名前をつけて実行してみよう。
$ python bosvm.py
...(省略)...
{'max_val': 0.9797297297297298, 'max_params': {'C': 31.622294778743424, 'gamma': 0.011103966665153005}}
上記から、最も汎化性能が高くなるのは C が 31.6 前後で gamma が 0.01 前後のとき、ということが分かった。
同時に、次のようなグラフが得られる。
これは、各ハイパーパラメータの値ごとに汎化性能を対数スケールの散布図で表したもの。
ハイパーパラメータによる精度の違いは元の数値ではぶっちゃけ微々たるものなので、数値は 0 ~ 1 に正規化してある。
また、汎化性能の高低は色で表現している。
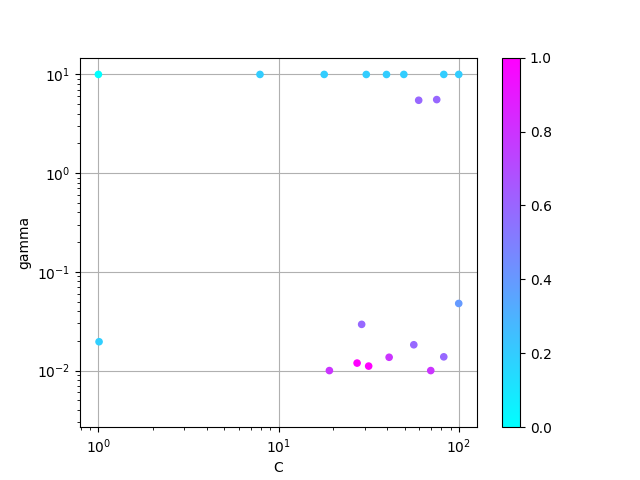
参考までに、グリッドサーチで調べた結果も以下に示す。

上記のグラフを描くのに使ったソースコードは次の通り。
import numpy as np
from sklearn import datasets
from sklearn.svm import SVC
from sklearn.model_selection import GridSearchCV
from matplotlib import pyplot as plt
from matplotlib import cm
def main():
dataset = datasets.load_iris()
X, y = dataset.data, dataset.target
clf = SVC(kernel='rbf')
params = {
'C': [1e+0, 1e+1, 1e+2],
'gamma': [1e-2, 1e-1, 1e+0, 1e+1],
}
grid_search = GridSearchCV(clf,
param_grid=params,
cv=3)
grid_search.fit(X, y)
score_and_params = np.array([(params['C'], params['gamma'], mean) for params, mean, _ in grid_search.grid_scores_])
scores = score_and_params[:, 2]
standard_scores = (scores - scores.min()) / (scores.max() - scores.min())
sc = plt.scatter(score_and_params[:, 0], score_and_params[:, 1], c=standard_scores, s=20, zorder=10, cmap=cm.cool)
plt.colorbar(sc)
plt.xlabel('C')
plt.xscale('log')
plt.ylabel('gamma')
plt.yscale('log')
plt.grid()
plt.show()
if __name__ == '__main__':
main()
まとめ
- ベイズ最適化を使うと未知の関数の出力が最大あるいは最小になる場所を見つけることができる
- 機械学習モデルにおけるハイパーパラメータの選択は未知の関数の出力を最大化あるいは最小化する問題と捉えられる
- つまり、機械学習モデルのハイパーパラメータ選びにベイズ最適化を適用できる場合がある